서 론
재료 및 방법
공시재료 및 RNA 추출
바이러스 동정을 위한 RT-PCR, In vitro transcription 및 표준시료 제작
SYBR Green I 및 TaqMan probe용 프라이머, 프로브 제작
Real-time PCR
Real-time PCR과 single or multiplex RT-PCR간의 비교
결과 및 고찰
적 요
서 론
2017년 국내 과수묘목 생산액은 약 618억원이며, 이는 국내 종자산업 규모의 10.4%에 달한다(KOSIS, 2018). 과수묘목 산업은 그 규모에 비해 굉장히 노동 및 공간 집약적이며 묘목생산에 많은 시간이 소요된다는 단점을 가지고 있다. 게다가 대부분의 묘목은 병원성 검정을 전혀 하지 않고 유통되고 있으며, 사과묘목의 경우 20~40%가 바이러스에 감염되어 있는 것으로 조사되었다(Chae, 2016). 바이러스에 감염된 사과는 당도가 2~5°Bx 정도 떨어지며 수세 또한 낮아지는 것으로 보고되었다(Chae, 2016). 이렇듯 사과 바이러스는 과실품질을 떨어뜨려 상품성을 낮추어 생산농가의 소득저하를 야기한다. 따라서 이러한 바이러스를 효과적으로 통제하기 위해서는 정확하고 정밀한 검정법의 개발이 필수적이며, 이를 통해 감염주의 유통을 막는 것이 근원적인 해결책이라 할 수 있다.
과수작물의 바이러스 검정법의 개발은 장비의 발달과 함께 발전해 왔다. 제일 먼저 시도된 생물학적 검정은 바이러스에 감수성인 지표식물에 바이러스 즙액을 접종시켜 그 병징을 보는 방식이었다(Kim et al., 2004). 그러나 이 방법은 시간이 많이 소요되고 민감도가 떨어졌으며, 어떠한 바이러스인지 특정 지을 수 없는 한계가 있었다(Kim et al., 2004). 그 다음은 전자현미경으로 직접 바이러스를 관찰하는 방법이었다. 이 방법은 바이러스를 눈으로 직접 볼 수 있어 바이러스의 외부 특성에 따른 분류를 가능하게 하였으나, 검역과 같은 대량의 검정체계에는 적합치 않았다(Heo et al., 2019). 바이러스 검정에 널리 활용되었던 방법 중의 하나인 ELISA (enzyme-linked immunosorbent assay) 검정법은 특정 바이러스에 대한 항체를 이용한 검사법으로, 바이러스 특이적인 검정과 정량이 가능하고 대량의 진단이 용이한 방법이다(Maliogka et al., 2018). 그러나 항체의 질이 떨어지고 공급이 제한되면서 그 대안으로 분자수준의 진단법이 제시되었다.
RNA 바이러스를 검정하기 위해 RT-PCR (reverse transcription-polymerase chain reaction) 검사법이 적용되기 시작했는데, 이 방법은 뛰어난 민감도와 정확성 그리고 편이성으로 인해 널리 활용되었다(Kumar et al., 2014). 또한 바이러스 여러 종을 한번에 검정하는 multiplex RT-PCR 검사법이 개발되어 바이러스 연구나 식물검역 등과 같은 분야의 대량검정에 이용되었다(Menzel et al., 2002). Real-time PCR 장비가 개발되면서 바이러스 검정 분야에도 활용되어 여러 진단법이 개발되었다(Mackay, 2004). 최근에는 Apple stem grooving virus (ASGV), Apple stem pitting virus (ASPV) 및 Apple mosaic virus (ApMV)를 동시에 진단할 수 있는 multiplex real-time PCR법이 개발되어 보고되었다(Malandraki et al., 2017).
그러나 여러 종의 바이러스를 한번에 검정하는 복합진단법은 프라이머간의 간섭과 증폭과정 중의 경쟁 등으로 인하여 민감도가 낮아지거나 위음성 결과(false negative result)를 낼 수 있는 문제점을 갖고 있다. 이는 real-time PCR 장비에서도 마찬가지이기 때문에 우리는 ASGV, ASPV 및 ApMV를 개별적으로 진단할 수 있는 singleplex real-time PCR 진단법을 개발하고자 하였다. 또한 바이러스 연구 및 검역현장에 바로 활용될 수 있도록 TaqMan probe 뿐만 아니라 SYBR Green I을 이용한 검정법을 개발해 비교하였다. 또한 현장평가를 통해 본 실험에서 개발된 검정법을 기존의 RT-PCR 결과와 비교하여 그 효율성과 민감도를 입증하고자 하였다.
재료 및 방법
공시재료 및 RNA 추출
국립원예특작과학원 사과시험장에서 유지되고 있는 ASGV, ASPV 및 ApMV 감염주(홍로, 감홍, 후지)에서 잎 시료를 채취하였다. 시료는 모수의 여러 부분에서 무작위로 채취한 후, RNA 추출 전까지 -80°C에 보관하였다. 잎 디스크를 무작위로 채취한 후, 액체질소를 이용해 마쇄하였다. 총 RNA는 CTAB method (Chang et al., 1993)를 약간 변용하여 추출하였다. 추출된 RNA는 침전시킨 후, 침전물을 diethylpyrocarbonate-treated water에 녹였다. 1% (w/v) agarose gel에 전기영동하여 RNA의 질을 평가하고, Nano Drop ND-1000 spectrophotometer (Thermo Scientific, Waltham, MA, USA)으로 정량하였다.
바이러스 동정을 위한 RT-PCR, In vitro transcription 및 표준시료 제작
각 바이러스의 외피단백질 지역의 일부를 동정하기 위해서, conventional RT-PCR을 수행하였다(Hassan et al., 2006). 실험에 사용한 프라이머는 Hassan et al. (2006)의 보고를 따랐으며, 길이는 ASGV의 경우에는 273 bp, ASPV는 370 bp, ApMV는 450 bp였다. 각 바이러스의 밴드는 젤에서 잘라내어 DNA를 추출하였다.
PCR증폭산물을 pGEM-T Easy vectors (Promega, Madison, WI, USA)에 삽입한 후, Escherichia coli strain JM110의 competent cell에 형질전환시켰다. 삽입된 DNA는 colony PCR로 확인한 후, 염기서열 분석을 수행하였다. 그런 다음 High Pure Plasmid Isolation Kit (Roche, Mannheim, Germany)를 이용해 plasmid DNA를 추출하고 제한효소 SalI (37°C, 3시간 30분)으로 절단하였다. 삽입방향에 따라 RiboMax Large Scale RNA Production Systems SP6 and T7 Kit (Promega)를 활용해 RNA를 전사시켰다: 2 µl linearized recombinant plasmid DNA (1 µg)를 주형으로 RNase inhibitor와 T7(또는 SP6) RNA polymerase를 37°C에서 2시간 방치, 그런 다음 plasmid DNA를 RNase-free DNase I으로 37°C에 30분 그리고 65°C에 10분동안 처리하였다. 각 바이러스의 nanogram당 ssRNA의 copy수 계산은 다음 식에 따라 계산하였다: copies per nanogram = (NA x A) / (n x mw), NA는 Avogadro 상수, A는 standard의 양, n은 standard의 nucleotide 길이, 그리고 mw는 nucleotide의 molecular weight.
SYBR Green I 및 TaqMan probe용 프라이머, 프로브 제작
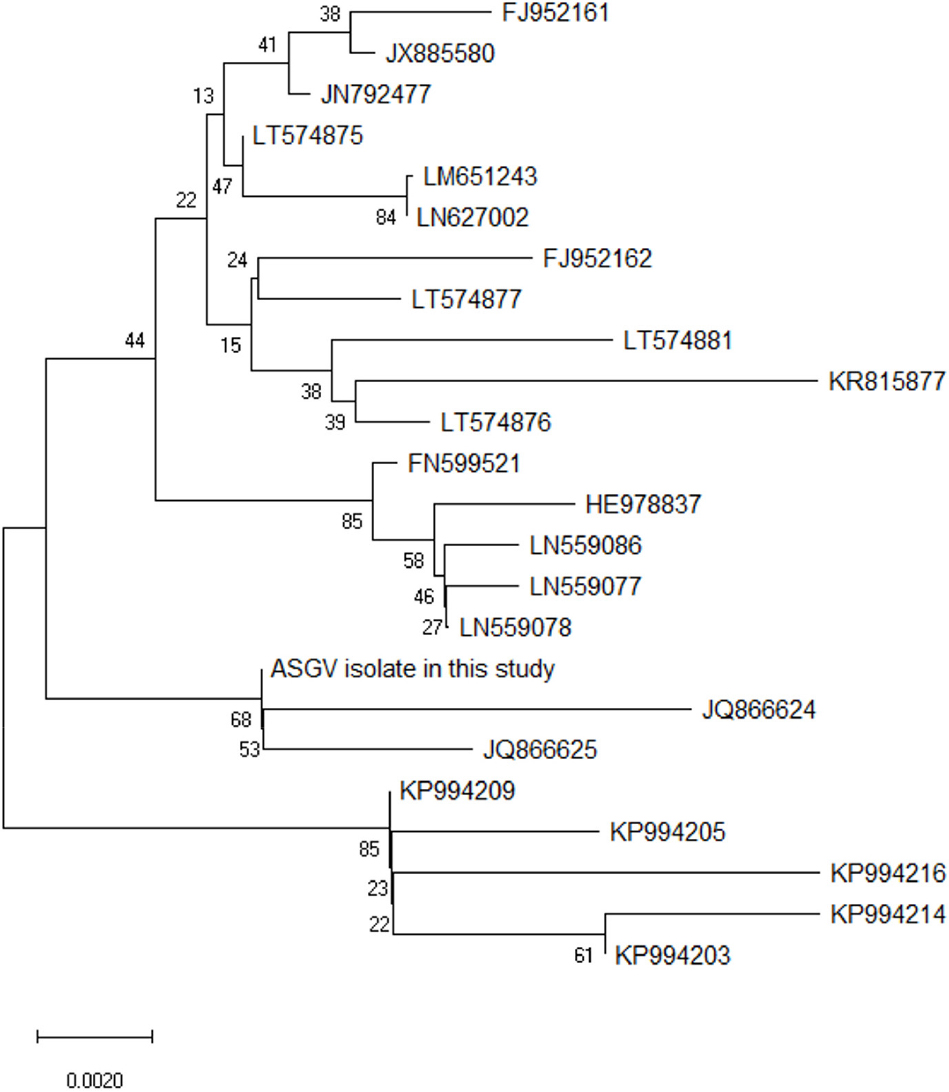
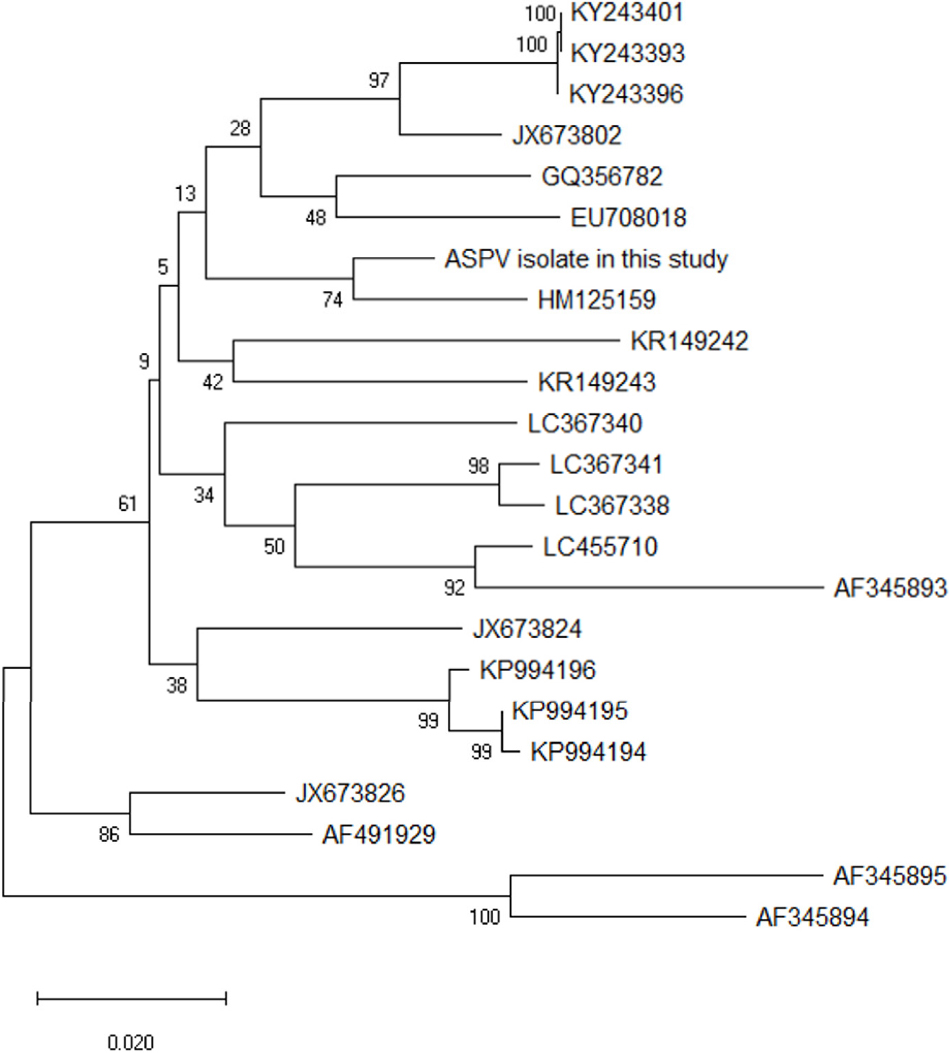
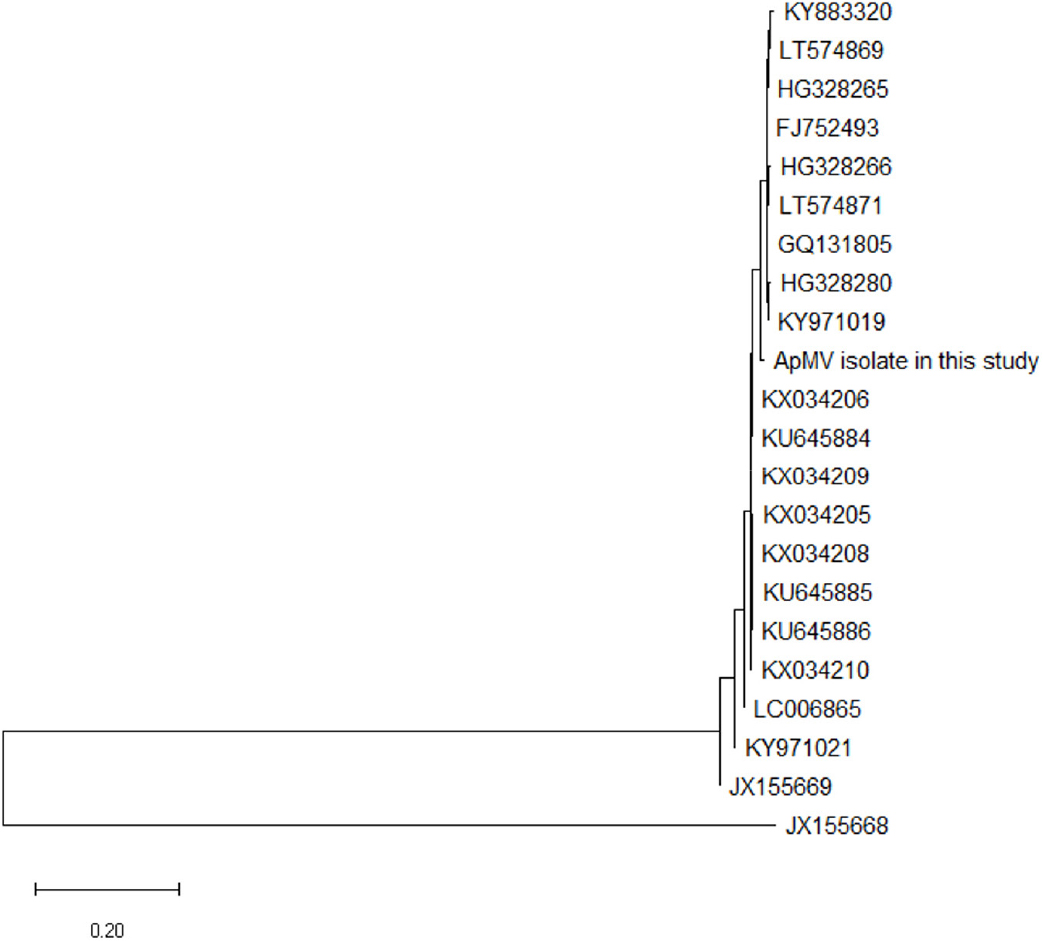
국내분리주 바이러스(Korean isolates) 및 Genbank에 등록된 유사유전자(homologous genes)를 대상으로 하여 계통도를 작성하였다(Fig. 1 for AGSV, Fig. 2 for ASPV and Fig. 3 for ApMV). Clustal W 방법으로 정렬하였고, 국내분리주와 유사유전자 간의 유전적 거리는 maximum composite likelihood model을 기반으로 한 neighbor-joining 방법으로 MEGA X (Kumar et al., 2018) 프로그램을 통해 계산하였다. 본 실험에서는 국내분리주의 염기서열을 바탕으로 유사유전자와 상동성이 높은 외피단백질 지역을 대상으로 Primer Express software version 2.0(Applied Biosystems, Foster City, CA, USA)를 이용해 프라이머와 프로브를 디자인하였다. TaqMan probe는 5’-말단에 6-carboxyfluorescein (FAM) reporter dye를, 3’-말단에는 6-carboxytetramethylrhodamine (TAMRA) fluorescent quencher를 붙여 제작하였다. SYBR Green I 및 TaqMan probe용 프라이머와 프로브의 염기서열 정보는 Table 1에 정리하였다.
Table 1.
The primers and probes of real-time PCR assay based on SYBR Green I dye and TaqMan probe for detection of ASGV, ASPV and ApMV apple viruses.
Real-time PCR
cDNA는 High Capacity cDNA Reverse Transcription Kit (Applied Biosystems)를 이용해 합성하였다. SYBR Green I에 기반한 real-time PCR (ABI 7500 Fast Real-time PCR Detection System)은 SYBR Premix EX Taq Kit (Takara, Kusatsu, Shiga, Japan)를 사용하였으며, PCR 조건은 다음과 같다: 95°C에서 enzyme 활성화 30초, 다시 95°C에서 denaturation 5초, 60°C에서 annealing과 extension을 30초의 조건으로 40회 반복하여 PCR 증폭을 수행한다. TaqMan probe를 이용한 real-time PCR은 TaqMan Universal PCR Master Mix Kit (Applied Biosystems)를 사용하였다. 최종 볼륨은 20 µl로 주형 DNA 2 µl, TaqMan probe 1 µl 그리고 10 µl의 TaqMan Universal Master Mix가 포함되었다. 반응조건은 제조사의 방법에 따라 수행하였다.
Real-time PCR과 single or multiplex RT-PCR간의 비교
82개의 사과 잎 시료를 대상으로 본 실험을 통해 개발된 real-time PCR 검정법과 기존의 singleplex 및 multiplex RT- PCR 간의 결과를 비교하였다(Table 2). 모든 시료는 경북 군위에 소재하고 있는 사과시험장 유전자원 포장에서 주요재배품종 및 대목품종을 대상으로 시료를 채취하였다. Singleplex RT-PCR은 다음의 프라이머를 사용하였다: 5’-TGGAAGA CGTGCTTCAAC-3’ (ASGV-F), 5’-TGTCCTTCAGTAC GAAAAAG-3’ (ASGV-R), 5’-GAGGCAGTATTGTGCCTT- 3’ (ASPV-F), 5’-CGGATGCGGTACATCTG-3’ (ASPV-R), 5’-CGAACCCGAATAAGGGTA-3’ (ApMV-F), 5’-GGTG GTAACTCACTCGTT-3’ (ApMV-R). PCR조건은 48°C에서 1시간 동안 방치 및 94°C에서 2분 동안 enzyme활성화한 후, 94°C에서 다시 1분 동안 denaturation, 51°C에서 annealing을 30초 그리고 72°C에서 elongation 단계를 1분간 총 40회 반복한 다음 final extension을 72°C에서 5분동안 수행하였다. PCR 증폭 후 agarose gel에 전기영동하였다. Multiplex RT-PCR의 경우, 상기 동정을 하기 위해 기술하였던 것과 마찬가지로 Menzel et al. (2002)의 방법에 따라 수행하였다. 모든 증폭산물은 시퀀싱을 통해 염기서열을 분석한 후, 비특이적 증폭을 배재하기 위해 BLAST (http://blast.ncbi. nlm.nih.gov/) 를 통해 확인하였다.
Table 2.
Detection of apple viruses from field samples by real-time PCR with TaqMan probe and conventional RT-PCR. Positive samples are indicated by cycle thresholds (Ct) and (+), for real-time PCR and conventional RT-PCR, respectively.
결과 및 고찰
각 바이러스의 증폭산물은 pGEM T-easy vector를 이용해 동정한 후, 염기서열을 분석하였다. 각 바이러스의 염기서열에 대한 blast를 수행한 결과, ASGV는 accession number LN901432 (ASGV CP gene for coat protein, e-value 3e-126), ASPV는 KP994189 (ASPV isolate Verbnae SMX coat protein gene, 1e-150), ApMV는 LC006865 (ApMV CP gene for coat protein, 0.0)와 상동성이 높았다. In vitro transcription 결과, ASGV는 1.46 x 1012, ASPV는 1.12 x 1012 그리고 ApMV는 1.56 x 1012 RNA copies/µl로 계산되었다. 이후 10배씩 희석시킨 표준시료를 작성한 후, ABI 7500 Fast 장비를 이용한 모든 실험을 3반복으로 수행하였다.
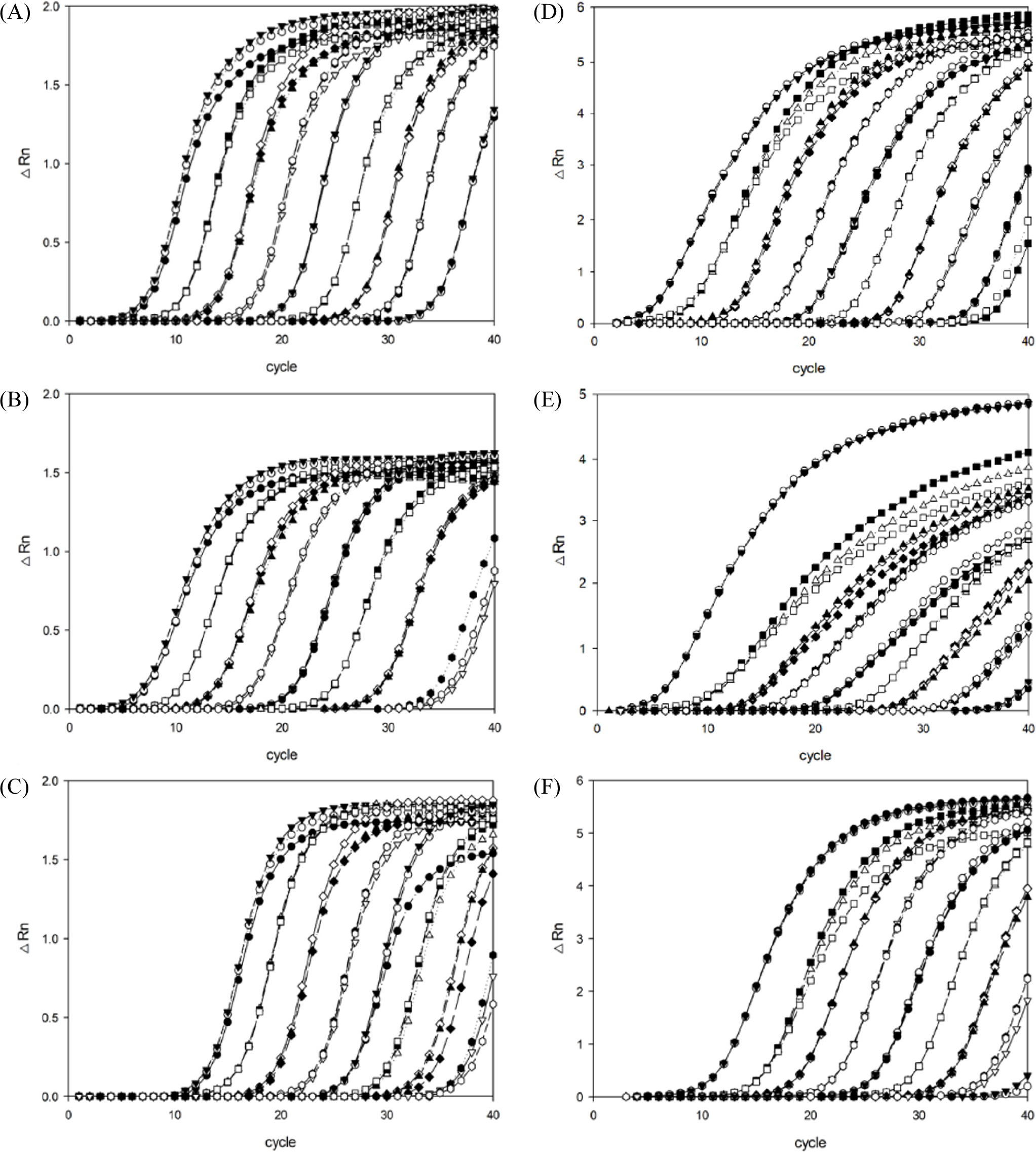
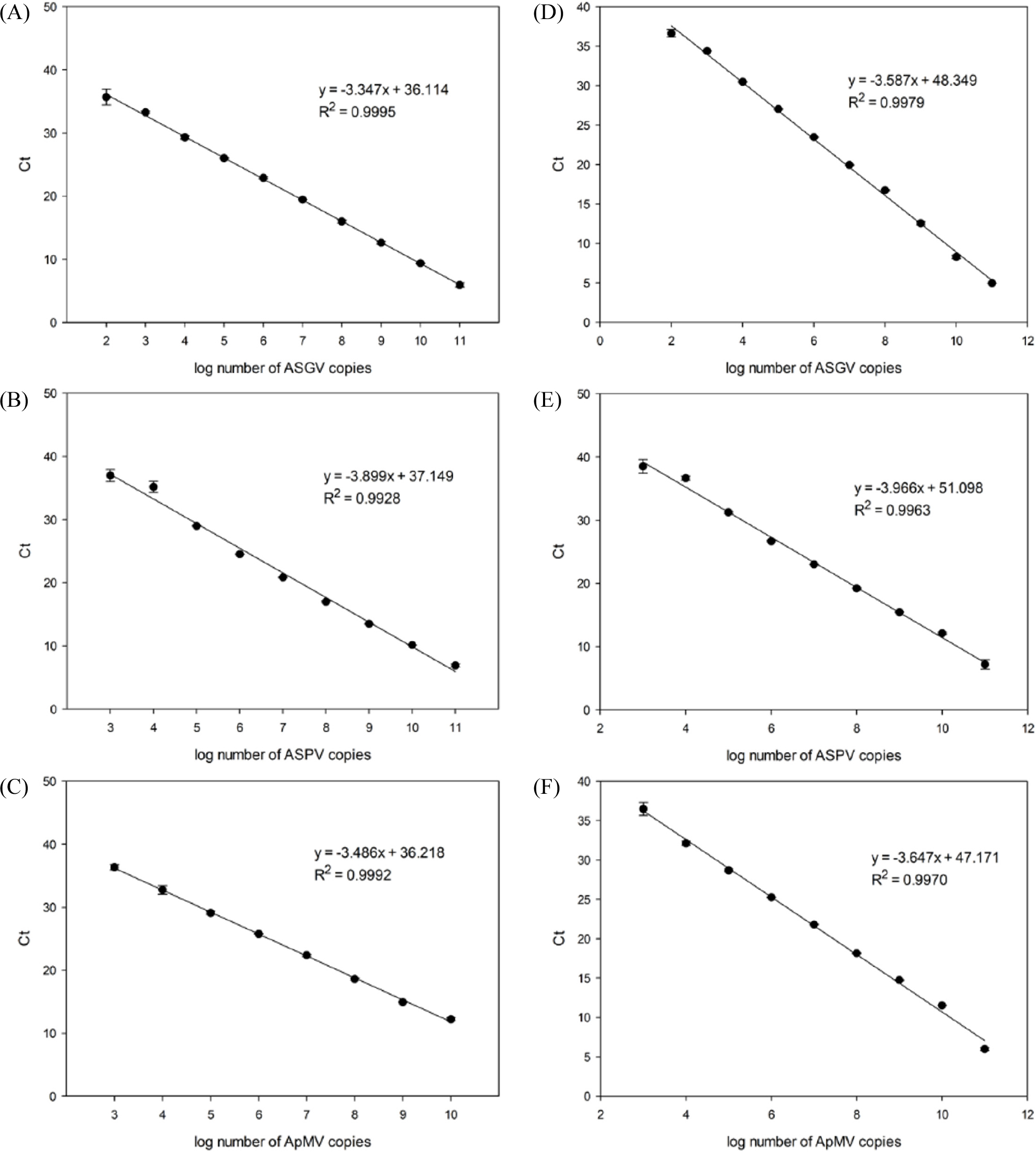
Fig. 4는 각 바이러스의 10배 희석한 표준시료를 대상으로 real-time PCR을 수행한 증폭곡선(amplification plot)으로, SYBR Green I dye를 이용한 실험(Fig. 4A~C)과 TaqMan probe를 이용한 실험(Fig. 4D~F)을 나타내고 있다. 10배 희석시킨 표준시료 간에는 약 2.5 cycles정도의 균일한 간격이 유지되고 있어 희석배수 간의 차이를 정확히 보여주었다. Fig. 5에서 보면, 각 바이러스의 10배 희석 표준시료에 대한 real-time PCR을 수행하여 얻은 threshold cycles (Ct) value를 Y축에, 각 바이러스 주형의 copy수의 log값을 X축에 표시하였다. 10배 단위 농도로 희석된 각 바이러스를 주형으로 하여 real-time PCR을 수행한 결과를 바탕으로, 주형의 농도에 따른 Ct 값의 회귀직선(linear regression)의 방정식과 회귀상수(regression coefficient, R2)를 구하였다(Fig. 5). 각 바이러스의 limit of detection (LOD)는 dye의 종류와 상관없이 마찬가지로 ASGV의 경우에는 102까지, ASPV와 ApMV는 103까지 측정이 가능하였다. 그러나 농도가 103까지 낮아질수록 Ct값과 검량선(standard curve) 간의 차이가 발생했으며, 또한 반복 간의 오차도 켜져서 limit of quantification (LOQ)도 103으로 설정하였다(ASGV는 102까지 정량이 가능하였다). 검량선 또한 형광물질에 따라 달리 작성하였는데(Fig. 5), 이 검량선을 이용하여 미지의 시료에 대한 감염 바이러스의 농도를 정확하게 추정할 수 있다.
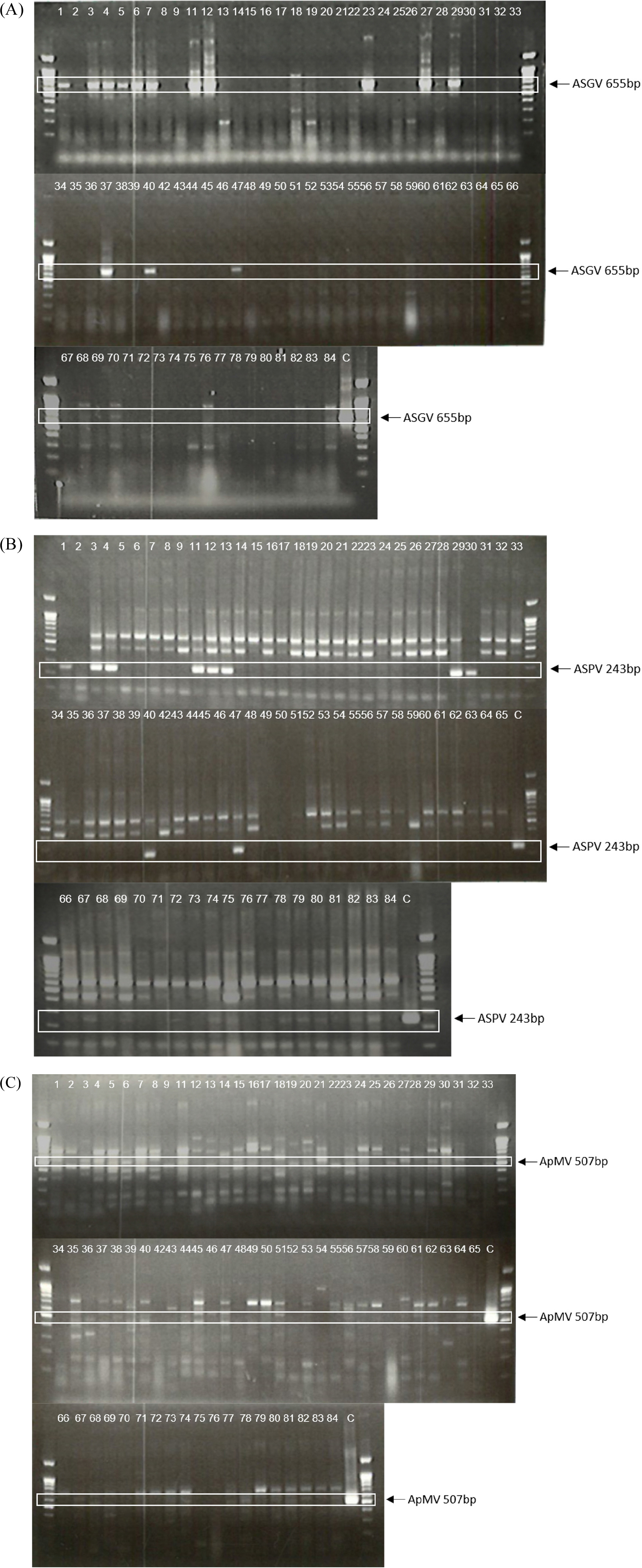
사과 유전자원 포장에서 유지되고 있는 사과 품종 및 대목품종의 82개 시료를 대상으로 본 실험에서 개발한 TaqMan probe 기반의 real-time PCR 검정법과 기존의 RT-PCR 검사법을 비교하였다(Table 2). RT-PCR은 각 바이러스를 개별적으로 검사한 singleplex RT-PCR과 두 종의 바이러스를 같이 진단하는 multiplex RT-PCR을 같이 수행하여 비교하였다. 82개 시료 중에서 real-time PCR을 통해 ASGV는 28.0% (23/82), ASPV는 29.3% (24/82), ApMV는 6.1% (5/82)가 양성으로 나왔다. 반면에 singleplex RT-PCR에서는 ASGV는 25.6% (21/82), ASPV는 25.6% (21/82), ApMV는 2.4% (2/82)만이 양성으로 판정되었다(Table 2 & Fig. 6). Multiplex RT-PCR에서는 singleplex RT-PCR보다 더 낮은 양성판정률을 보여주었다(Table 2 & Fig. 7). 이 결과에서 보듯 multiplex RT-PCR은 동시에 여러 바이러스를 검정할 수 있는 장점이 있으나, 정밀하게 진단하는데 제한이 있는 것으로 판단된다. 이는 multiplex PCR의 경우, 프라이머 간의 간섭, PCR증폭과정 상의 경쟁 등으로 인해 민감도가 떨어져 위음성 결과를 초래할 가능성이 있다. 본 실험을 통해 개발된 real-time PCR검정법은 RT-PCR검정법으로 검출하지 못할 정도로 낮은 농도의 시료까지도 검출이 가능하였다.
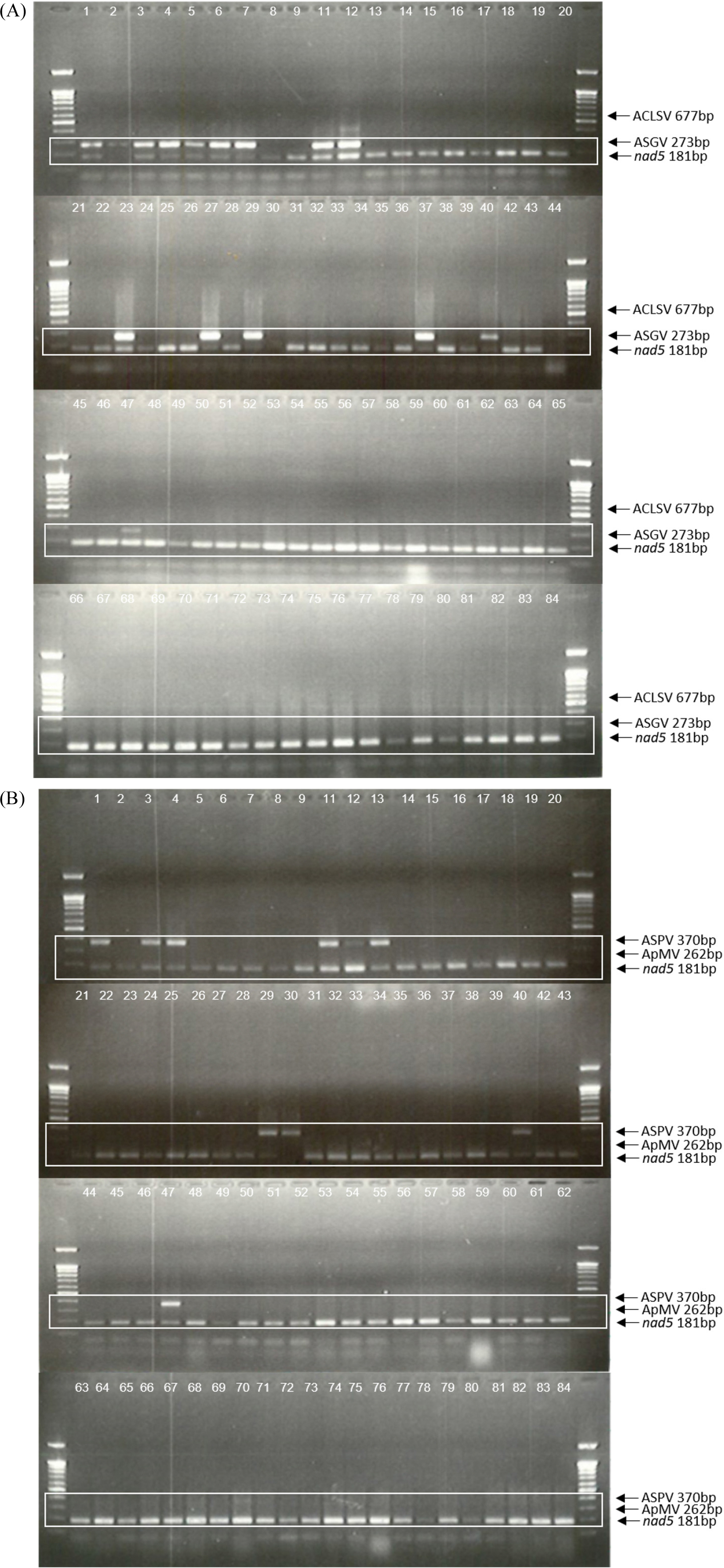
Fig. 7
The results of multiplex RT-PCR: (A) ACLSV+ASGV; (B) ASPV+ApMV. An internal control gene, nad5 (accession no. D37958) from apples, was used to guarantee the PCR amplification based on RNA, not DNA (Menzel et al., 2002).
사과 바이러스 중 ASGV, ASPV 및 ApMV는 경제적으로 큰 피해를 주는 병원이다(Magome et al., 1997; Nickel & Fajardo, 2014; Winkowska et al., 2016). 이 바이러스들은 미세섬유 구조의 입자 형태이며 ssRNA genome을 갖는다(Nickel & Fajardo, 2014). ASGV와 ASPV는 일반적으로 사과 나무에 별다른 병징을 나타내지 않는다고 하였으나(Nemeth, 1986), ASGV는 접목부위에 괴사를 야기하는 것으로 보고되었다(Nickel et al., 1999). ASPV는 접목전염을 통해서 과실이 쪼개지는 증상(crack)이 있다고 알려졌다(James et al., 2013). ApMV는 ASGV와 ASPV와 달리 그 병징이 바로 눈으로 확인가능한데, 잎에 밝은 노란색의 모자이크 패턴이 발생한다(Nickel & Fajardo, 2014). 또한 위 바이러스들은 단독감염보다는 여러 개의 바이러스가 같이 감염되는 복합감염이 더 일반적이다. 위 바이러스에 복합감염된 사과나무는 심각할 정도로 수세가 약화되며 과실생산량도 급격히 감소한다(Chae, 2016). 이러한 바이러스들의 복합감염의 주 원인은 대목의 생산 과정에 있다. 모든 사과품종은 접목묘 형태로 생산, 유통되는데, 대목은 주로 대목모수를 이용한 취목(ground layering)에 의해 생산된다. 이때 대목 모수가 바이러스에 감염되어 있는 경우가 많아 여기서 무성번식으로 생산된 모든 대목이 바이러스에 감염된 상태에서 품종접수로 접목을 하게 된다. 대부분의 접목묘가 2~3년생으로 유통되는데, 유묘기에는 바이러스의 병징이 전혀 나타나지 않고 과실이 생산되는 성목기가 되어서야 비로소 병징이 발현되기 때문에 농가의 경제적 피해가 크다. 따라서 바이러스 감염묘의 생산, 유통을 방지할 수 있는 근원적인 대책이 필요하며, 그것이 바로 정확하고 민감한 검정법의 개발에 있다고 할 수 있다. 이러한 검정법을 대목생산부터 접목묘 생산에까지 적용하여 감염주를 제거하고 무독묘를 유지시킨다면 감염묘로 인한 경제적 피해를 사전에 차단할 수 있을 것이다. 또한 검역현장과 같이 대량의 바이러스 진단이 필요한 곳에서는 본 실험을 통해 개발된 SYBR Green I기반의 real-time PCR검사법이 적은 비용에 대량검사가 가능하여 높은 효율을 낼 수 있을 것으로 기대된다. 또한 바이러스 연구분야에서는 정밀하게 바이러스를 정량하는 방법이 필수적이므로 TaqMan probe를 이용한 real-time PCR검정법이 활용될 수 있을 것이다.
적 요
본 연구는 사과 바이러스 ASGV, ASPV 및 ApMV를 각각 정밀하게 진단하고자 SYBR Green I 및 TaqMan probe, 두 종류의 다른 chemical dye를 사용하여 quantitative real-time PCR 검정법을 개발하고자 하였다.
1. 사과 바이러스 ASGV, ASPV 및 ApMV의 국내분리주를 바탕으로 하여 cloning 및 in vitro transcription을 수행해 10배 희석단위 표준시료를 제작하였다. 각 바이러스에 대한 SYBR Green I용 프라이머와 TaqMan probe용 프라이머 및 프로브를 디자인하였다.
2. 상기 제작된 프라이머와 프로브를 이용해 표준시료를 대상으로 real-time PCR을 수행하여 각 바이러스의 증폭곡선과 검량선을 구할 수 있었다. Real-time PCR 결과, SYBR Green I기반의 검정법은 TaqMan probe기반의 검정법 못지 않은 결과를 보여주었으며, 적은 비용에 대량 검정이 요구되는 곳에 효과적으로 응용될 수 있을 것이다.
3. 현장평가를 본 실험에서 개발된 TaqMan probe기반의 real-time PCR검정법과 기존의 RT-PCR검정법과 비교분석하였다. 그 결과 real-time PCR 검정법은 singleplex 및 multiplex RT-PCR보다 더 민감하고 정확한 결과를 내어 RT-PCR로 검출할 수 없는 농도까지 검정할 수 있음을 입증하였다.
4. 본 실험에서 개발한 real-time PCR검정법은 검역현장과 같은 대량의 검사가 요구되는 곳에서는 SYBR Green I기반의 검정법을, 바이러스 연구분야에서는 세밀한 정량이 가능한 TaqMan probe 방식의 검정법이 활용될 것으로 기대한다.