서 론
재료 및 방법
시험재료 및 재배방법
입형 관련 표현형 조사
입형 관련 유전자형 분석
통계분석
결과 및 고찰
입형 관련 표현형 분석
입형 관련 대립유전자형 분석
생태형별 대립유전자 조합에 따른 입형 특성
자포니카 품종의 입형 관련 대립유전자 계보 분석
적 요
서 론
벼는 가장 중요한 식량작물 중 하나로 전세계 인구의 50% 이상이 주식으로 이용하고 있으며 전세계 칼로리 섭취량의 20%를 담당하고 있다(Huang et al., 2012). 최근 전세계 인구 증가와 경지면적의 감소, 빠르게 변화하는 시장 속에서 품종의 수량 증진, 품질 향상 그리고 기존 품종과 다른 차별성 있는 품종 개발 전략이 필요하다(Mo et al., 2020). 종자의 입형 특성은 수량과 품질에 중요한 역할을 할 뿐만 아니라 인종, 문화 성별 및 연령에 따라 낟알의 크기와 형태를 선호하는 특성이 다르다(Ngangkham et al., 2018; Park et al., 2018). 입형은 상품의 시장가치와 소비에 영향을 주며 주로 우리나라, 일본, 중국 북부에서는 둥근 형태의 낟알 특성을 선호하지만, 유럽, 동남아시아, 중국 남부 등에서는 낟알이 길이가 긴 것을 선호한다(Calingacion et al., 2014).
입형을 구분하는 국제적인 기준은 없지만 우리나라에서는 현미 길이를 기준으로 5.00 mm 이하 단립종(short), 5.01- 5.50 mm 중단립종(medium-short), 5.51-6.00 mm 중립종(medium), 6.01-7.00 mm 장립종(long), 7.01 mm 이상 초장립종(extra-long)으로 구분하며(RDA, 2012), 국제 식물 신품종 보호 동맹(International Union for the Protection of New Varieties of Plants; UPOV) 기준을 따라 현미의 형태를 장폭비 1.5 이하 원형(round), 1.50-1.99 단원형(semi-round), 2.00-2.49 중원형(half spindle-shaped), 2.50-2.99 장원형(spindle-shaped), 3.00 이상 세장형(long spindle-shaped)으로 구분한다(KSVS, 2005). 재배벼는 주로 자포니카와 인디카의 생태형으로 구분되는데 자포니카는 낟알의 길이가 짧고 형태가 둥근 단립종의 단원형 입형특성을 가지는 것이 많고, 인디카벼는 낟알의 길이가 길고 너비가 얇은 장립종의 세장형 입형특성을 나타내는 것이 많다(Juliano, 1979). 우리나라 벼 품종들은 예로부터 차지고 끈기가 있는 자포니카 쌀을 좋아하여 자포니카 위주로 품종 개발 및 쌀 산업 구조가 발달되었지만 지금까지 개발된 품종 대부분 중단립종 단원형의 매우 협소한 입형 특성을 가지고 있어 수량성 증대와 새로운 품질에 대한 소비자의 요구 충족, 쌀 산업 발전을 위한 사회적 변화에 대응하는데 한계가 있다(Park et al., 2018).
입형은 여러 유전자가 관여하여 조절되는 양적형질로 지금까지 약 400여개 이상의 많은 양적형질유전자좌(quantitative trait loci: QTL)가 탐색되었다(Huang et al., 2013). 낟알의 크기와 형태 관련 형질에 영향을 주는 중요 유전자와 QTL 중 클로닝되어 그 위치와 기능이 밝혀진 QTL은 GW2, GS3, qGL3/qGL3.1, qSW5/GW5 (이하 qSW5), GS5, TGW6, GW7, GW8 등이 있다. GW2는 염색체 2번에 위치하고 있으며 유전자 염기 서열의 결실로 인해 단백질 생성이 조기 종결됨으로써 유전자의 기능을 상실하게 되어 낟알의 너비, 무게와 수량을 증가시킨다(Song et al., 2007). GS3는 염색체 3번에 위치하며 낟알 길이와 무게에 주동으로 작용하는 유전자로 관련 유전자 말단에 기관 크기 조절(OSR; organ size regulation) 도메인의 기능상실이나 결실, 염기서열의 돌연변이로 인해 낟알이 길어지게 된다(Fan et al., 2006). qGL3.1은 염색체 3번에 있는 QTL로 이삭에서 단백질 인산화에 관여하여 종실의 길이와 수량을 조절한다(Qi et al., 2012). qSW5와 GW5는 5번 염색체에서 낟알의 너비와 무게에 관여하는 동일 주동 QTL로 밝혀졌으며 대부분의 자포니카 벼에서 발견되는 qSW5 염기서열의 1212-bp 결실로 인해 낟알의 너비가 길어지고 무게가 증가하게 된다(Park et al., 2018; Shomura et al., 2008; Weng et al., 2008). GS5는 염색체 5번에서 양의조절작용(positive regulator)을 하여 낟알의 크기에 영향을 준다(Li et al., 2011). TGW6 유전자는 IAA-glucose hydrolase를 암호화하여 세포수와 종자 길이를 조절하지만 인도 재래벼인 Kasalath에서 발견된 TGW6는 기능을 상실하여 낟알 길이가 길어지고 낟알의 무게가 증가하게 된다(Ishimaru et al., 2013). GW7 염색체 7번에 있는 QTL로 유전자 발현 시 낟알에서 종 방향으로 세포 분열이 증가하고 횡 방향으로는 세포 분열이 감소하게 되어 가느다란 낟알 생성에 영향을 준다(Wang et al., 2015). GW8 염색체 8번에 있는 QTL로 OsSPL16와 동일한 기능을 한다고 밝혀졌으며 GW8이 발현되면 세포 분화와 등숙에 영향을 줄 뿐만 아니라 낟알의 너비를 길어지게 한다(Wang et al., 2012).
이처럼 입형 특성은 벼의 수량과 품질에 중요한 역할을 하기 때문에 많은 연구가 진행되었지만 지금까지 국내에서 육성된 벼 품종에 대한 유전자형 구분은 이루어진 바가 없다. 본 연구는 자포니카 벼의 수량성 향상과 입형 다양화, 품질 경쟁력 강화를 위해 기존에 보고된 유전자(GW2, GS3, qGL3, qSW5/GS5, TGW6, GW7 and GW8)를 이용하여 국내에서 육성된 벼 품종들의 입형 관련 형질과 유전자형과의 관계를 분석함으로써 육종사업에 활용할 수 있는 자료를 제공하고자 수행하였다.
재료 및 방법
시험재료 및 재배방법
농촌진흥청 국립식량과학원에서 육성된 벼 272품종을 시험에 이용하였다. 시험재료를 국립식량과학원 벼 시험포장에서 2017-2018년 2년간 보통기 재배방법으로 4월 30일 파종하여 5월 30일에 재식거리 30 × 15 cm로 주당 1본씩 조당 26주를 이앙하였다. 시비량은 N-P2O5-K2O를 90-45-57 kg/ha으로 질소는 기비 : 분얼비 : 수비를 50 : 20 : 30 비율로 분시하였고, 인산은 전량 기비로, 칼륨은 기비 : 수비를 70 : 30 비율로 분시하였다. 기타 재배관리는 농촌진흥청 표준 재배법에 준하여 실시하였다.
입형 관련 표현형 조사
육성품종의 출수 후 약 50일경을 수확기로 하여 성숙이 제대로 이루어진 평균개체를 수확하여 자연 건조 후 탈곡하여 정조를 정선하였다. 제현기(SY88-TH, Ssang Yong Machine Ind., Korea)를 이용하여 제영된 현미를 이용하여 입형 관련 형질을 조사하였다. 등숙이 충실이 이루어진 현미를 선별하여 길이, 너비, 두께를 캘리퍼스(Caliper CD-15CP, Mitutoyo Corp., Japan)를 이용하여 5립씩 조사하고 너비에 대한 길이의 비율로 장폭비를 계산하였다. 선별된 현미 100립에 대한 무게를 3반복으로 측정하여 평균 값을 천립중으로 환산하였다. 육성품종의 입형 관련 형질 값은 2017-2018년 2년간의 평균 조사 값을 이용하였다.
입형 관련 유전자형 분석
육성품종의 입형 관련 유전자형 분석을 수행하였다. Genomic DNA 추출은 BioSprint 96 (Qiagen Co., Düren, Germany)을 이용하였다. 샘플을 TissueLyser II (Qiagen Co., Düren, Germany)로 마쇄한 후 BioSprint 96 DNA Plant Kit (Qiagen Co., Düren, Germany)를 이용하여 DNA를 추출하였다. 입형 관련 유전자 GW2, GS3, qGL3, LGY3, qSW5, GS5, TGW6, GW7, GW8를 탐지하는 분자표지를 이용하여 유전자형을 분석하였다. 분자표지는 기존의 연구결과에서 적용되었던 GW2-Hpa I (GW2 대상), GS3-Pst I (GS3), Xj24 (qGL3), OsMADS1-INDEL (LGY3/OsMADS1), N1212del (qSW5), GS5-03SNP (GS5), TGW6-1d (TGW6), GW7-INDEL (GW7), GW8-INDEL (GW8)를 이용하였다(Kim et al., 2016; Park et al., 2018; Wu et al., 2018).
PCR은 10 ng의 DNA와 2X Multiplex PCR Smart Mix (Solgent, Korea)를 이용하여 T-1 Thermoblock (Biometra, Germany)에서 수행하였다. PCR 반응은 GW2-Hpa I, GS3-Pst I, qGL3는 94°C에서 4분간 초기 변성 후 94°C 45초, 55°C 45초, 72°C 1분간 35회 반복하고 72°C에서 5분간 반응하였다. N1212del는 94°C에서 5분간 초기 변성 후 94°C 45초, 65°C 45초, 72°C 2분간 30회 반복하고 72°C에서 10분간 반응하였다. GW2와 GS3의 증폭된 PCR 산물은 제한효소 Hpa I, Pst I를 처리하고, qSW5와 qGL3 제한효소 처리 없이 전기영동하였다. SYBR safe DNA gel stain (Invitron)으로 염색된 2% agarose gel을 이용하여 전기영동을 수행하였고, UV transilluminator (MiniBIS Pro, DNR Bio-Imaging Systems Ltd., Jerusalem, Israel)를 활용하여 유전자형을 판정하였다.
통계분석
통계분석은 R (Version 3.6.3, The R Foundation for Statistical Computing Platform)을 이용하였다. 육성품종의 생태형, 입형 관련 유전자의 대립유전자형과 유전자형 조합에 따른 형질의 평균 등 기술통계, t-test, Ducan’s Multiple Range Test (DMRT)를 이용한 평균간 비교를 기본 및 agricolae 패키지를 이용하여 수행하였다(de Mendiburu & de Mendiburu, 2019). 입형 관련 형질 값에 대한 boxplot과 violin plot을 ggplot2 패키지를 이용하여 작성하였다. 육성품종의 입형 관련 표현형에 대한 산포도, 히스토그램, 형질의 상관계수를 psych, 주성분분석을 ggfortify, K-means 군집분석을 factoextra 패키지를 이용하여 분석하였다(Kassambara & Mundt, 2017; Revelle, 2017; Tang et al., 2016).
결과 및 고찰
입형 관련 표현형 분석
국내 육성 벼 272품종의 입형 관련 형질에 대한 표현형을 분석하였다(Fig. 1). 육성품종을 생태형에 따라 자포니카와 통일형으로 구분하였고, 자포니카 생태형 중 입형 특성이 다른 것으로 파악된 흑미 품종을 따로 구분하여 자포니카, 흑미, 통일형 세 개의 생태형으로 분류하여 입형 관련 특성을 비교 분석하였다(Fig. 1A). 생태형별 품종수는 자포니카 225개(82.7%), 흑미 14개(5.2%), 통일형 33개(12.1%)로 자포니카 품종이 대부분을 차지하였다. 현미 길이는 통일형(5.89 mm)과 흑미(5.85 mm)가 자포니카(5.20 mm)에 비해 길었으며, 너비와 두께는 자포니카(2.90, 2.06 mm), 흑미(2.70, 1.93 mm), 통일형(2.56, 1.85 mm) 순으로 넓고 두꺼웠다. 장폭비는 통일형(2.31), 흑미(2.17), 자포니카(1.79) 순으로 커 자포니카에 비해 흑미와 통일형이 갸름했으며, 천립중은 자포니카(21.0 g), 통일형(20.3 g), 흑미(19.2 g)순으로 무거워 자포니카가 통일형과 흑미에 비해 무거웠다.
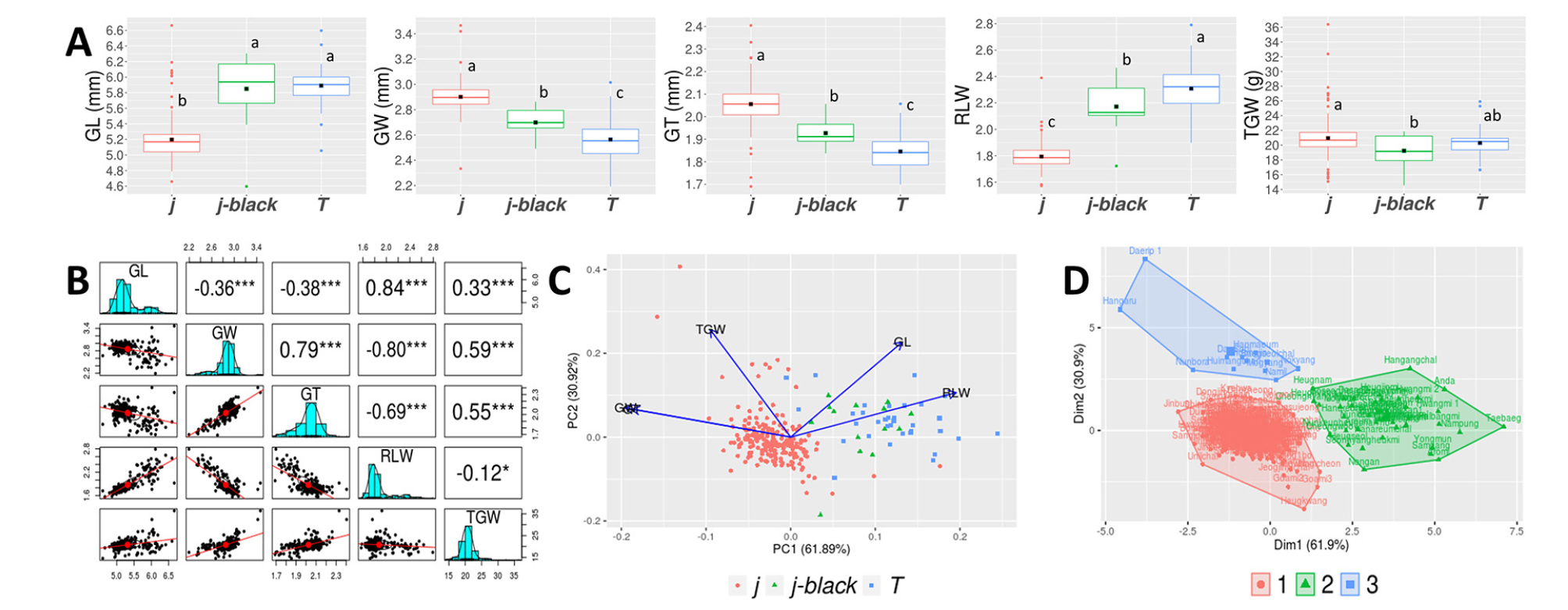
Fig. 1.
Phenotypic characterization of grain-related traits of Korean rice varieties. Boxplots of grain-related traits according to ecotypes (A). Black rectangles indicate the means of grain-related traits. GL: gain length, GW: grain width, GT: grain thickness, RLW: length to width ratio, TGW: 1,000-grain weight. j: japonica rice, j-black: japonica black rice, T: Tongil-type rice ecotype. Different letters above the bars indicate significant differences among ecotypes. Scatterplot matrices, histograms, and correlations for grain-related traits (B). Red circles, red lines, and black ellipses represent the means, linear regression lines, and correlation ellipses, respectively. Principle component analysis among grain-related traits (C). PC1: principle component 1, PC2: principle component 2. K-means cluster analysis of Korean rice varieties by grain-related traits (D). Dim1: dimension 1, Dim2: dimension 2. 1: cluster 1, 2: cluster 2, 3: cluster 3.
국내 육성 벼 품종(이하 육성품종)의 입형 특성은 이상치 값을 나타내는 품종을 포함하여 단립종-장립종(4.60-6.66 mm), 단원형-장원형(1.57-2.79)의 입형 분포를 보였으며, 생태형에 따라 평균적으로 자포니카는 중단립종에 단원형, 흑미와 통일형은 중립종에 중원형 입형 특성을 나타냈다(Fig. 1A). 육성품종의 입형 관련 형질 분포를 살펴보면 길이와 장폭비는 왼쪽으로 치우친 정규분포, 너비와 두께는 오른쪽에 치우친 정규분포를 나타냈으며 천립중은 형질 값이 상당히 큰 품종이 존재하는 분포를 나타냈다(Fig. 1B). 현미 길이는 장폭비와 상관계수 0.84로 높은 정의 상관관계를 나타냈으며, 너비와 두께와는 -0.36, -0.38로 부의 상관관계를 나타냈다(Fig. 1B). 너비와 두께는 상관계수 0.79로 높은 정의 상관관계를 나타냈으며, 장폭비와 너비는 -0.80, 두께는 -0.69으로 높은 부의 상관관계를 나타냈다. 천립중에 대해서는 너비(상관계수 0.59), 두께(0.55), 길이(0.33) 순으로 상관관계가 높았으며, 장폭비는 천립중과 상관관계가 낮았다. 이러한 결과는 521개 벼 유전자원을 대상으로 5개 입형 관련 형질들의 상관관계를 분석한 결과와 비슷한 경향이었다(Zhong et al., 2019).
주성분분석을 통해 입형 관련 형질과 육성품종의 구조적 관계를 파악하였다(Fig. 1C). 주성분 1과 2는 전체 형질 변이의 61.89%와 30.92%를 설명하여 이들 2개 주성분으로 육성품종의 입형 관련 형질 변이 대부분을 설명할 수 있었다. 주성분 1을 기준으로 현미 너비와 두께는 같은 방향성과 효과를 나타내며 천립중과 함께 장폭비, 길이와 다른 방향성을 나타냈고, 주성분 2를 기준으로는 모든 형질이 같은 방향성을 보였으나 다른 형질들에 비해 천립중과 길이가 영향을 많이 미치는 것으로 판단되었다. 주성분 분포도에서 자포니카 품종은 대부분 근접되어 분포하고 있으나 입형 관련 형질 값이 이상치를 보인 몇몇 품종들은 확연하게 다른 위치에 분포하고 있다. 통일형과 흑미 품종은 주로 주성분 1을 기준으로 장폭비와 길이가 위치하는 방향에 분포하여 자포니카와 구분되는 것을 확인할 수 있었다.
K-means 군집분석을 수행하여 입형 관련 표현형에 따라 육성품종들의 그룹을 분류하였다(Fig. 1D). 군집의 수가 2, 3, 4, 5개일 때 전체 형질 변이를 설명하는 정도가 47.7, 62.7, 70.3, 73.6%로 2개에서 3개로 군집이 변경될 때 상대적으로 설명하는 정도가 커지기 때문에 군집의 수를 3개로 정하여 분석을 수행하였다. 군집 1, 2, 3은 각각 육성품종 217, 43, 12개를 포함하였는데, 군집 1은 흑미 품종 ‘흑광’, ‘흑수정’, 통일형 품종 ‘남천’, ‘녹우’를 제외한 213개 품종(98.2%)이 자포니카 생태형이었다. 군집 2는 43개 품종 중 군집 1에 포함된 품종을 제외한 흑미 생태형 85.7%에 해당하는 흑미 12품종, 통일형 생태형 87.9%에 해당하는 통일형 29품종 등 흑미와 통일형 41품종(95.3%)과 자포니카 품종 ‘농안’, ‘아로미’로 구성되어 있으며, 군집 3에는 12개 품종 중 ‘대립벼1호’, ‘신동진’, ‘남일’ 등 자포니카 10품종(83.3%)과 통일형 품종 ‘녹양’, ‘목양’이 포함되었다. K-means 군집분석을 위해 이용된 R 패키지 factoextra는 주성분분석을 기반으로 변수의 차원을 축소하여 새로운 변수를 생성하게 되기 때문에 차원 1과 2는 주성분분석의 주성분 1과 2와 같으며 각 품종의 배치도 주성분분석 행렬도와 같은 위치에 있게 된다(Kassambara & Mundt, 2017). 주성분분석의 형질 분포와 군집분석의 그룹을 대응해 보면 그룹 1은 현미 너비와 두께, 그룹 2 장폭비와 길이, 그룹 3은 천립중이 영향이 많이 미치는 것으로 판단되었다(Fig. 1C and D). 자포니카 품종 대부분으로 구성된 그룹 1, 흑미와 통일형 품종 위주로 구성된 그룹 2, 주로 자포니카 품종이 포함된 그룹 3의 현미 길이, 너비, 두께, 장폭비, 천립중은 그룹 1이 5.18 mm, 2.93 mm, 2.09 mm, 1.79, 21.5 g, 그룹 2가 5.91 mm, 2.57 mm, 1.85 mm, 2.31, 19.7 g, 그룹 3이 6.03 mm, 3.09 mm, 2.13 mm, 1.95, 27.5 g으로, 천립중은 그룹 3, 1, 2 순으로 무거웠으며 그룹 1은 중단립종에 단원형, 그룹 2는 중립종에 중원형, 그룹3은 장립종에 단원형 입형 특성을 나타냈다.
입형 관련 대립유전자형 분석
입형 관련 유전자 GW2, GS3, qGL3, LGY3/OsMADS1(이하 LGY3), qSW5/GW5(이하 qSW5), GS5, TGW6, GW7, GW8를 탐지하는 분자표지를 이용하여 육성품종의 입형 관련 대립유전자형을 분석하였다. 동일한 대립유전자형을 나타낸 LGY3에 제외한 8개 유전자에 대해서 단일 유전자의 대립유전자형에 따른 입형 관련 형질 값을 비교 분석하였다(Table 1, Fig. 2).
Table 1.
Comparison of grain-shape traits of Korean rice varieties according to allele types of eight genes associated with grain shape.
| Gene (reference) | Allele type (effect) | Traits related to grain shape | |||||
| no. (%) | GLy (mm) | GW (mm) | GT (mm) | RLW | TGW (g) | ||
| GW2 (Song et al., 2007) | gw2WY3 (+)z | 3 (1.1) | 6.10**x | 3.32** | 2.33** | 1.84ns | 31.8** |
| GW2FAZ1 (N) | 269 (98.9) | 5.31 | 2.84 | 2.02 | 1.88 | 20.7 | |
| GS3 (Fan et al., 2006) | gs3A-allele (+) | 23 (8.5) | 6.01aw | 2.78b | 1.95b | 2.18b | 21.7a |
| GS3B-allele (M) | 12 (4.4) | 5.80b | 2.49c | 1.82c | 2.34a | 19.3b | |
| GS3C-allele (N) | 237 (87.1) | 5.22c | 2.87a | 2.04a | 1.82c | 20.8ab | |
| qGL3 (Zhang et al., 2012) | qgl3N411 (+) | 30 (11.0) | 5.89** | 2.53** | 1.83** | 2.34** | 19.9* |
| qGL39311 (N) | 242 (89.0) | 5.24 | 2.89 | 2.05 | 1.82 | 20.9 | |
| qSW5 (Shomura et al., 2008) | qsw5_NNipponbare (+) | 259 (95.2) | 5.29b | 2.86a | 2.03a | 1.86c | 20.8a |
| qsw5_IIndica II (M) | 6 (2.2) | 5.76a | 2.73a | 1.96a | 2.12b | 21.2a | |
| qSW5Kasalath (N) | 7 (2.6) | 5.91a | 2.49b | 1.79b | 2.38a | 19.7a | |
| GS5 (Li et al., 2011) | gs5H94 (N) | 259 (95.2) | 5.28** | 2.86** | 2.03** | 1.85** | 20.8ns |
| GS5Zhensan97 (+) | 13 (4.8) | 5.97 | 2.61 | 1.89 | 2.31 | 20.8 | |
| TGW6 (Ishimaru et al., 2013) | tgw6Kasalath (+) | 1 (0.4) | 5.94ns | 2.71ns | 1.88ns | 2.20ns | 21.2ns |
| TGW6Nipponbare (N) | 271 (99.6) | 5.31 | 2.85 | 2.02 | 1.87 | 20.8 | |
| GW7 (Wang, 2015) | gw7Nipponbare (+) | 118 (43.4) | 5.28ns | 2.89** | 2.05** | 1.83** | 21.2** |
| GW79311 (N) | 154 (56.6) | 5.34 | 2.82 | 2.00 | 1.91 | 20.4 | |
| GW8 (Wang, 2012) | gw89311 (N) | 31 (11.4) | 5.89** | 2.54** | 1.83** | 2.32** | 20.1ns |
| GW8Nipponbare (+) | 241 (88.6) | 5.24 | 2.89 | 2.05 | 1.82 | 20.9 | |
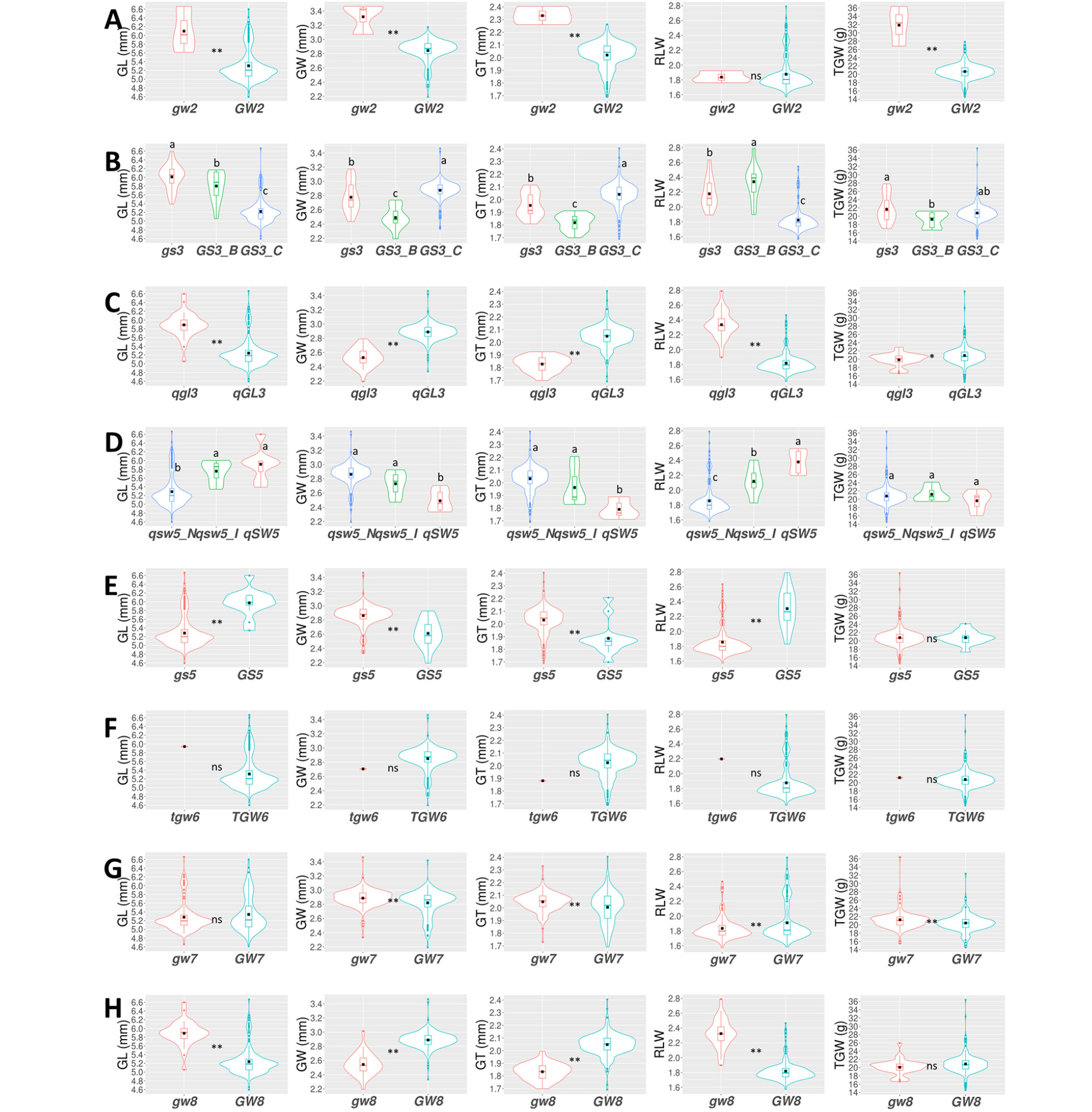
Fig. 2.
Violin plots of grain-related traits according to allele types of GW2 (A), GS3 (B), qGL3 (C), qSW5 (D), GS5 (E), TGW6 (F), GW7 (G), and GW8 (H). Black rectangles indicate the means of grain-related traits. GL: grain length, GW: grain width, GT: grain thickness, RLW: length to width ratio, TGW: 1,000-grain weight. Abbreviation of allele types are listed in Table 1. ns, *, and ** mean no significant, significant at p<0.05, and p<0.01 by t-test, respectively. Means with same letters in a column are not significant at p<0.05 (ANOVA followed by DMRT).
GW2 유전자는 E3 ubiquitin ligase 활성을 가지는 RING- type 단백질을 부호화하는데, GW2의 기능 상실은 왕겨의 세포수 증가와 등숙속도의 향상을 가져와 종실의 너비, 무게와 수량을 증가시킨다(Song et al., 2007). 육성품종 중 종실크기가 커지는 것으로 알려진 대립유전자 gw2를 가지고 있는 것은 자포니카 ‘대립벼1호’, ‘눈보라’, ‘한가루’ 3품종이었다. 대립유전자 gw2형 품종군은 GW2형과 현미 장폭비는 비슷하였으나 길이, 너비, 두께가 크고 천립중이 무거웠다(Fig. 2A). 육성품종 중 gw2 대립유전자 빈도는 1.1%로 매우 낮았고 자포니카에서만 확인되었는데, 이러한 결과는 벼를 소비하는 28개국의 215개 유전자원을 통한 대립유전자형 분석에서 gw2를 보유한 자원은 ‘대립벼1호’, ‘Hokuriku 130’, ‘Oochikara’ 3품종 밖에 안되는 등 gw2 대립유전자의 존재는 드물다는 결과와 일치하였다(Lee et al., 2015).
GS3 유전자는 최초로 클로닝되고 기능이 밝혀진 유전자로 종실의 길이와 무게에 주동으로 작용하며 N 말단에 기관 크기 조절(organ size regulation: OSR) 도메인이 존재하는데 이 부위의 기능상실이나 결실은 긴 종실의 특성을 가져온다(Fan et al., 2006; Mao et al., 2010). 초기 GS3 대립유전자는 엑손 2에 존재하는 A와 C의 단일염기다형성(SNP)에 의한 2개의 대립유전자형이 존재하고, A-allele (gs3)은 시스테인 코돈(TGC)을 종결코돈(TGA)으로 넌센스 돌연변이를 일으켜 GS3 유전자 기능상실에 의하여 종실 길이와 무게가 증가하는 것으로 알려져 있다(Fan et al., 2009). 이후 다른 연구결과에서 A, C-alelle 이외에 첫번째 인트론에 45 bp의 염기서열 삽입에 의한 다른 대립유전자형인 B- allele이 존재하며, B-allele은 종실 길이와 장폭비가 A와 C-allele의 중간에 속한다고 하였다(Lee et al., 2015). 육성품종의 GS3 대립유전자형은 A, B, C-allele과 같은 gs3, GS3_B, GS3_C 세 개를 나타냈는데, 현미 길이는 gs3형 품종군의 가장 길었고 GS3_B, GS3_C형 순으로 길어 이전 연구결과와 같은 경향이었다(Fig. 2B). GS3_C 대립유전자를 가진 품종이 237개(87.1%)로 대부분을 차지하였고, gs3 보유 23품종, GS3_B 보유 12품종이었다(Table 1). GS3_B 대립유전자형 12품종은 모두 통일형 생태형이었으며, gs3를 보유한 23품종은 ‘신동진’ 등 자포니카 6품종, ‘흑광’을 제외한 흑미 13품종, ‘한강찰’ 등 통일형 4품종이었다.
qGL3는 종실 길이에 관여하는 주동 양적형질 유전자좌로 OsPPKL1 추정 단백질을 부호화하며, 기능상실이 일어날 경우 낟알 길이가 길어지는 부의 조절 작용을 한다(Zhang et al., 2012). 육성품종 중 길이가 길어지는. 것으로 알려진 대립유전자 qgl3를 보유한 것은 30품종으로 모두 통일형 품종이었다. 흑미를 포함한 자포니카 생태형 239개 품종은 모두 qGL3 대립유전자를 가지고 있었으며, 통일형 33개 품종 중 qGL3를 가지고 있는 품종은 교배조합에 열대자포니카 계통이 포함된 사료용 3품종 ‘녹양’, ‘목양’, ‘녹우’로 qgl3은 통일형 생태형에 특이적인 대립유전자로 생각되었다. qgl3형 품종군은 qGL3형에 비해 현미 길이가 길고 너비는 좁아 장폭비가 컸고 두께가 얇고 천립중이 가벼웠다(Fig. 2C).
종실의 너비와 무게에 관여하는 유전자 qSW5는 벼의 순화 과정에 관련된 유전자로 온대 자포니카 생태형 대부분은 qSW5 유전자 염기서열의 1,212 bp가 결실되어 있는데 이는 유전자의 기능상실을 가져와 종실의 너비가 넓어지고 무게가 증가하게 된다(Shomura et al., 2008). qSW5 유전자는 염기서열 결실에 의해 세 개의 대립유전자형이 존재하는데 결실이 없고 유전자의 기능을 가지는 aus 생태형의 Kasalath형(qSW5), 1,212 bp, 950 bp가 결실되어 기능이 상실된 온대 자포니카 Nipponbare형(qsw5_N) 및 인디카 Indica-II형(qsw5_I)이 있다(Yan et al., 2011). 육성품종의 qSW5 대립유전자형도 qsw5_N, qsw5_I, qSW5 세 개가 존재하였는데, qsw5_N형 품종군이 259개(95.2%)로 대부분을 차지하였으며 qsw5_I와 qSW5형 품종군은 각각 6개와 7개였다(Table 1). 자포니카 생태형 중 ‘남일’, ‘새오대’, ‘아로미’ 3품종을 제외한 자포니카 222품종과 흑미 14품종 등 총 236개 품종(98.7%)이 qsw5_N형을 나타내 대부분을 차지하였으며, 이를 통해 qsw5_N는 우리나라 자포니카 품종의 입형을 대표하는 대립유전자로 판단되었다. 통일형 33품종 중 23품종(70.0%)도 qsw5_N 대립유전자를 보유하고 있었는데, 이는 인디카/자포니카 교배에 의해 육성된 통일형 품종의 기원과 자포니카와 같이 단원형을 선호하는 우리나라 시장 요구가 육종과정 중에 반영되어 통일형 품종 개발의 선발압으로 작용한 결과 qsw5_N을 보유한 다수의 품종이 존재하는 것으로 생각된다. 6개 품종이 포함된 qsw5_I형 품종군에는 ‘다산1호’, ‘목우’, ‘영우’, ‘청우’ 등 통일형 4품종과 ‘남일’, ‘새오대’ 등 자포니카 2품종이 속하였고, 7개 품종이 포함된 qSW5형 품종군에는 ‘한강찰’, ‘남풍’, ‘다산’, ‘팔방미’, ‘미면’, ‘한아름찰’ 등 통일형 6품종과 자포니카 생태형으로 ‘아로미’가 단독으로 포함되어 있었다. 기능이 상실된 qsw5_N과 qSW5_I형 품종군이 기능을 가지고 있는 qSW5형에 비해 현미 너비가 넓고 두께가 두꺼웠다(Fig. 2D).
GS5 유전자는 종실의 너비, 등숙, 무게에 정의 조절 작용(positive regulator)을 하는 유전자로 5번 염색체 단완에서 qSW5 유전자와 약 2 Mb 아래에 위치한다(Li et al., 2011). 육성품종은 GS5에 대해서 두 개의 대립유전자형을 나타냈으며, GS5형을 보유한 품종은 13개로 통일형 ‘용주’, ‘다산’, ‘목우’ 등 11품종과 자포니카 ‘남일’, ‘새오대’ 였다. 종실 크기가 증가하는 것으로 알려진 GS5형 품종군은 gs5형에 비해 현미 길이와 장폭비는 증가하였으나, 너비와 두께는 오히려 감소하였고 천립중은 통계적으로 유의한 차이가 없었다(Fig. 2E). 이러한 결과는 입형 특성에 다른 입형 관련 유전자의 상호작용인 상위성이 작용하였거나, GS5 대립유전자는 상대적으로 품종수가 적은 통일형 생태형에 치우쳐 분포하고 있기 때문인 것으로 판단된다. 이에 생태형별로 GS5 대립유전자형에 의한 입형 특성을 비교 분석하였다. 통일형 생태형에서 GS5형 품종군(n = 11)의 입형 특성은 6.03 mm, 2.56 mm, 1.84 mm, 2.37, 20.3 g이었고 gs5형 품종군(n = 22)이 5.82 mm, 2.57 mm, 1.85 mm, 2.28, 20.3 g로 GS5형이 현미 너비, 두께, 천립중은 비슷하나 길이와 장폭비를 증가시켰다. 또한, 자포니카 생태형에서 GS5 대립유전자를 보유한 ‘남일’과 ‘새오대’의 평균 현미 길이, 너비, 두께, 장폭비, 천립중은 5.63 mm, 2.90 mm, 2.15 mm, 1.94, 23.4 g으로 자포니카 생태형의 평균 입형 특성인 5.20 mm, 2.90 mm, 2.06 mm, 1.79, 21.0 g에 비해 너비는 비슷하나 길이, 두께, 장폭비가 증가하여 천립중이 무거운 특성을 볼 때 GS5 대립유전자는 종자 크기를 증가시키는 것으로 판단되었다. 특이한 점으로 GS5 유전자와 5번 염색체 단완에 연관되어 있는 qSW5 유전자의 qsw5_I 대립유전자를 가지고 있는 6품종(자포니카 ‘남일’, ‘새오대’, 통일형 ‘다산1호’, ‘목우’, ‘영우’, ‘청우’) 모두 GS5형을 보유하고 있었다.
TGW6 유전자는 하나의 엑손을 가지고 있으며 ‘Kasalath’와 같이 이 부위에서 1 bp의 염기서열 결실이 일어날 경우 유전자 기능이 상실되고 이로 인해 종실의 천립중이 증가한다(Ishimaru et al., 2013). 육성품종 중 통일형 생태형 ‘미면’에서만 ‘Kasalath’와 같은 기능상실 대립유전자 tgw6가 나타났는데, 이는 기능상실 대립유전자형 빈도가 매우 낮다는 이전의 연구결과와 일치하였다(Ishimaru et al., 2013; Kim et al., 2016). 육성품종에서 tgw6 대립유전자의 빈도가 낮아 입형 특성에 대한 통계적으로 유의한 차이가 없었으나, tgw6를 보유한 ‘미면’의 현미 천립중이 21.2 g으로 통일형 생태형 평균 20.8 g 보다 높은 편으로 tgw6에 의한 천립중 증가효과는 있을 것으로 판단되었다(Fig. 2F).
GW7 유전자의 발현량 증가는 종 방향으로 세포 분열이 증가하고 횡 방향으로 분열이 감소되어 가느다란 종실 형태를 가져온다(Wang et al., 2015). 육성품종의 gw7와 GW7 대립유전자 빈도는 전체품종에서 43.4% (118품종)와 56.6% (154품종), 자포니카에서 48.4% (109품종)와 51.6% (116품종), 흑미에서 50.0% (7품종)와 50.0% (7품종)으로 균일한 대립유전자형 분포를 보였다. 하지만 통일형 생태형에서는 ‘녹양’과 ‘녹우’ 2품종(6.1%)만 gw7 대립유전자를 보유하고 있고 나머지 33품종(93.9%)는 GW7를 가지고 있어 편이된 분포를 보였다. 종실 너비가 증가하는 것으로 알려진 gw7형 품종군이 GW7형에 비해서 너비, 두께, 천립중이 다소 커진 경향이었으며 장폭비는 줄어들었다(Fig. 2G).
GW8 유전자의 발현량 증가는 세포분열과 등숙을 촉진시켜 종실 너비와 수량을 증가시킨다(Wang et al., 2012). 종실 너비가 줄어드는 대립유전자 gw8를 보유한 품종은 31품종으로 모두 통일형 생태형 품종이었다. gw7 대립유전자와 마찬가지로 ‘녹양’과 ‘녹우’ 2품종만 너비가 증가하는 GW8를 보유하고 있었고 나머지 통일형 품종은 폭이 줄어드는 gw8형을 나타내 gw8은 통일형 생태형에 특이적인 대립유전자로 생각되었다. 종실 너비가 증가하는 것으로 알려진 GW8형 품종군이 gw8형에 비해서 현미 너비, 두께가 증가하였으나 길이가 짧아 천립중은 비슷한 수준이었다(Fig. 2F).
입형 관련 대립유전자형 분석을 통해 국내 육성 벼 품종 중 빈도수가 1.1%와 0.4%였던 gw2와 tgw6형은 매우 드문 대립유전자이며, 통일형 생태형에서만 존재하였던 qgl3와 gw8 대립유전자는 종 특이적인 대립유전자로 판단되었다. 생태형별 대립유전자형의 빈도나 입형 특성을 고려할 때 국내 소비의 대부분을 차지하는 자포니카 밥쌀용 품종의 중단립종 단원형의 입형을 대표하는 대립유전자형은 qsw5_N인 것으로 생각되었다. 따라서 gw8, qgl3와 qSW5 등의 대립유전자가 포함되도록 품종을 육성한다면 우리나라 자포니카 생태형 벼 품종들의 입형 다양화에 도움이 될 것으로 생각된다.
생태형별 대립유전자 조합에 따른 입형 특성
국내 육성 벼 품종의 생태형별 입형 관련 대립유전자 조합에 따른 입형 특성을 분석하였다. 생태형별 대립유전자 조합의 수는 자포니카 7개(Cj1~Cj7), 흑미 3개(Cj_b1~Cj_b3), 통일형 13개(CT1~CT13)로 자포니카에 비해 품종수가 적은 통일형 생태형이 더 다양한 조합수를 나타냈으며 생태형별 대립유전자 조합을 대표하는 품종에 대한 입형 관련 유전자형과 표현형은 Fig. 3과 같다. 자포니카 품종은 qGL3, TGW6, GW8 유전자에서는 대립유전자의 다형성이 없었으며, 대립유전자 조합 Cj1 (GW2-GS3_C-qGL3-qsw5_N-gs5-TGW6-gw7-GW8)과 Cj2 (GW2-GS3_C-qGL3-qsw5_N-gs5-TGW6-GW7-GW8)에 속하는 품종이 101개와 112개로 대부분을 차지하며 중단립종·단원형의 거의 비슷한 입형 특성을 나타내며 gw7 대립유전자형을 가진 Cj1이 GW7형의 Cj2보다 현미 천립중에서 다소 무거웠다(Table 2, Fig. 4A). Cj3 (GW2-GS3_C-qGL3-qSW5-gs5-TGW6-gw7-GW8)은 Cj1에서 qsw5_N이 qSW5로 치환된 조합으로 ‘아로미’ 1품종만 이에 속하였고 Cj1에 비해 현미 너비, 두께, 천립중이 감소한 반면 길이는 증가하여 장폭비 2.39로 자포니카 품종 중 유일하게 중원형 입형 특성을 나타냈다. Cj4 (GW2-GS3_ C-qGL3-qsw5_I-GS5-TGW6-GW7-GW8)는 Cj2에서 qsw5_ N-gs5가 qsw5_I-GS5로 치환된 조합으로 ‘남일’과 ‘새오대’ 2품종이 여기에 포함되며, Cj2에 비해 현미 길이, 장폭비, 천립중이 증가하였다. Cj5 (GW2-gs3-qGL3-qsw5_N-gs5-TGW6- gw7-GW8)은 Cj1에서 GS3_C가 gs로만 치환된 조합으로 ‘신동진’, ‘소비’, ‘한마음’, ‘다미’, ‘백설찰’, ‘희망찬’ 등 국내에서 이른바 중대립종으로 불리는 품종들이 포함되며, Cj1에 비해 현미 두께를 제외하고 길이, 너비, 장폭비, 천립중이 증가하여 장립종 및 중원형에 근접하였으며 무거운 입형 특성을 나타냈다. Cj6 (gw2-GS3_C-qGL3-qsw5_N-gs5- TGW6-GW7-GW8)와 Cj7 (gw2-GS3_C-qGL3-qsw5_N-gs5- TGW6-gw7-GW8)은 국내 육성품종 중 gw2를 보유한 조합으로 Cj2와 Cj1 조합에서 GW2가 gw2로 치환된 것으로 대립종으로 불리는 ‘눈보라’와 ‘한가루’(Cj6), ‘대립벼1호’(Cj7)가 여기에 속하며, 국내 육성품종 중 입형 관련 형질 값이 가장 큰 편이며 gw7 대립유전자형을 보유한 Cj7이 GW7형의 Cj6보다 형질 값이 더 컸다. 일반적으로 중단립종·단원형 입형으로 알려진 국내 육성 자포니카 품종을 감안할 때 중단립종·단원형을 나타내며 94.7%를 차지하는 대립유전자 조합 Cj1과 Cj2이 국내육성 자포니카 품종의 입형 특성을 대표되며, 여기에 gw2, gs3, qsw5, GS5 대립유전자가 도입됨으로써 다양성이 확대된 것으로 판단된다.
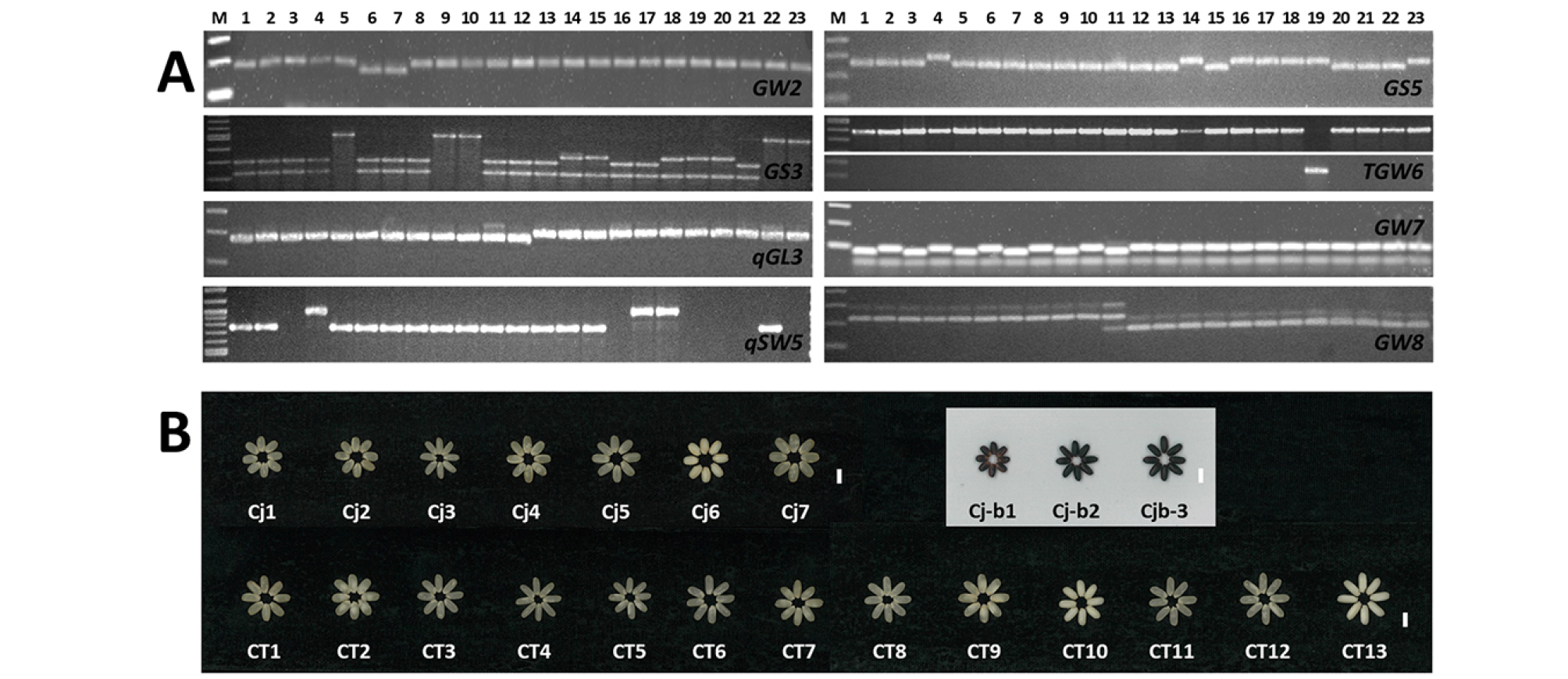
Fig. 3.
Genotype and phenotype of allele combinations. PCR analysis to confirm the allele types of varieties that represent the allele combinations of ecotypes using gene specific DNA markers. (A). M: size marker, 1: Ilmi (Cj1), 2: Ilpum (Cj2), 3: Aromi (Cj3), 4: Namil (Cj4), 5: Sindongjin (Cj5), 6: Nunbora (Cj6), 7: Daerip 1 (Cj7), 8: Heugkwang (Cj-b1), 9: Heugjinju (Cj-b2), 10: Boseogheugchal (Cjb-3), 11: Nokyang (CT1), 12: Mogyang (CT2), 13: Hanareum (CT3), 14: Taebaeg (CT4), 15: Samgang (CT5), 16: Dasan (CT6), 17: Mogwoo (CT7), 18: Dasan 1, 19: Mimyeon (CT19), 20: Hanareumchal (CT10), 21: Nampung (CT11), 22: Anda (CT12), 23: Hangangchal (CT13). Allele combinations are listed in Table 2. Image of brown rice of varieties that represent the allele combinations of ecotypes (B). Varieties and allele combinations are consistent with those mentioned above. White bars indicate 5 mm.
Table 2.
Grain-related traits of Korean rice varieties according to ecotypes and allele combinations.
zGL: grain length, GW: grain width, GT: grain thickness, RLW: length-to-width ratio, TGW: 1,000-grain weight
yCj1: GW2-GS3_C-qGL3-qsw5_N-gs5-TGW6-gw7-GW8, Cj2: GW2-GS3_C-qGL3-qsw5_N-gs5-TGW6-GW7-GW8, Cj3: GW2-GS3_C-qGL3-qSW5-gs5-TGW6-gw7-GW8, Cj4: GW2-GS3_C-qGL3-qsw5_I- GS5-TGW6-GW7-GW8, Cj5: GW2-gs3-qGL3-qsw5_N-gs5-TGW6-gw7-GW8, Cj6: gw2-GS3_C-qGL3-qsw5_N-gs5-TGW6-GW7-GW8, Cj7: gw2-GS3_C-qGL3-qsw5_N-gs5-TGW6-gw7-GW8.Abbreviation of allele types are listed in Table 1.
xCj-b1: GW2-GS3_C-qGL3-qsw5_N-gs5-TGW6-GW7-GW8, Cj-b2: GW2-gs3-qGL3-qsw5_N-gs5-TGW6-gw7-GW8, Cj-b3: GW2-gs3-qGL3-qsw5_N-gs5-TGW6-GW7-GW8
wCT1: GW2-GS3_C-qGL3-qsw5_N-gs5-TGW6-gw7-GW8, CT2: GW2-GS3_C-qGL3-qsw5_N-gs5-TGW6-GW7-gw8, CT3: GW2-GS3_C-qgl3-qsw5_N-gs5-TGW6-GW7-gw8, CT4: GW2-GS3_B-qgl3-qsw5_ N-GS5-TGW6-GW7-gw8, CT5: GW2-GS3_B-qgl3-qsw5_N-gs5-TGW6-GW7-gw8, CT6: GW2-GS3_C-qgl3-qSW5-GS5-TGW6-GW7-gw8, CT7: GW2-GS3_C-qgl3-qsw5_I-GS5-TGW6-GW7-gw8, CT8: GW2-GS3_B-qgl3-qsw5_I-GS5-TGW6-GW7-gw8, CT9: GW2-GS3_B-qgl3-qSW5-GS5-tgw6-GW7-gw8, CT10: GW2-GS3_B-qgl3-qSW5-gs5-TGW6-GW7-gw8, CT11: GW2-GS3_C-qgl3-qSW5-gs5-TGW6- GW7-gw8, CT12: GW2-gs3-qgl3-qsw5_N-gs5- TGW6-GW7-gw8, CT13: GW2-gs3-qgl3-qSW5-GS5-TGW6-GW7-gw8
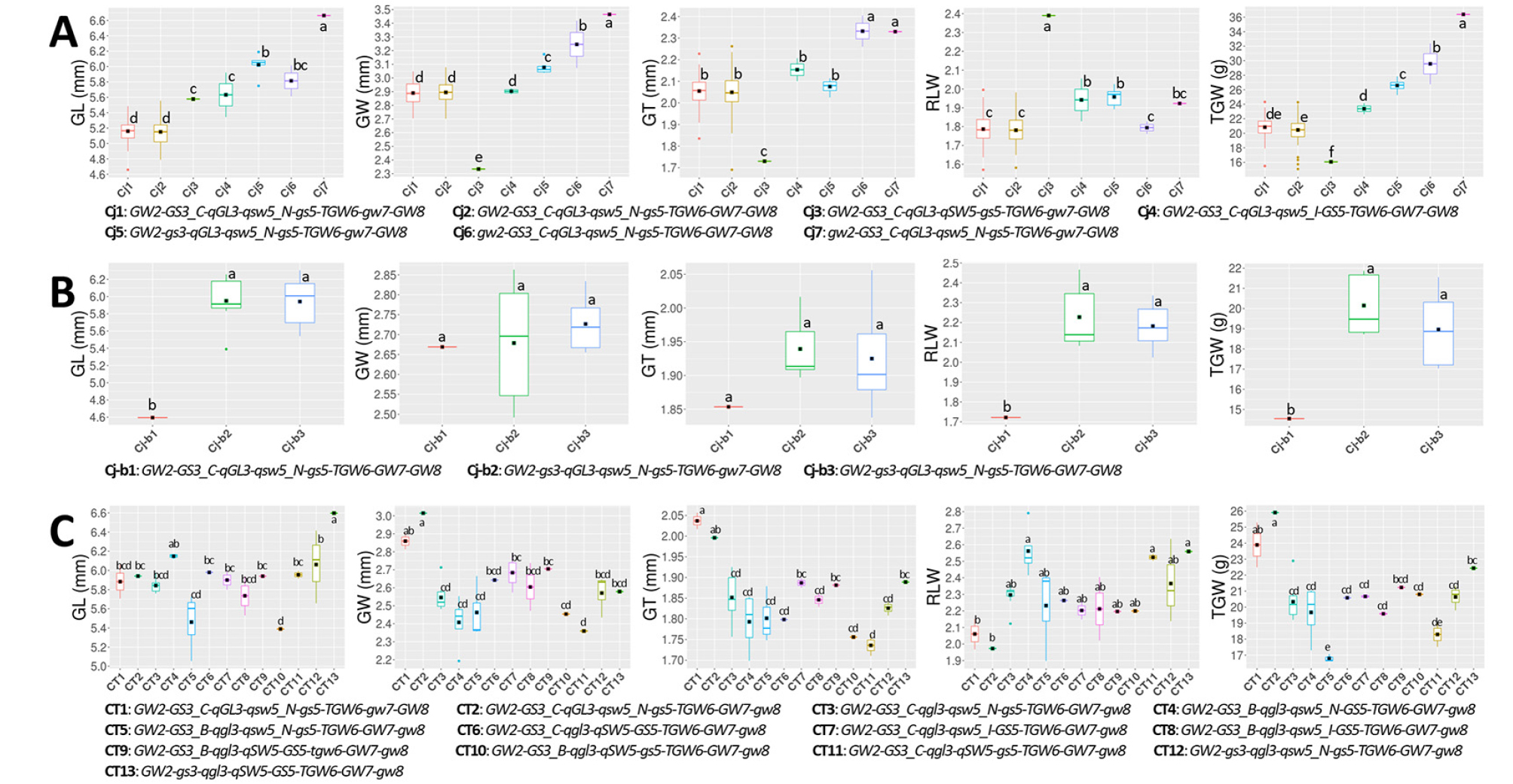
Fig. 4.
The effects of various allele combinations of japonica rice (A), japonica-black rice (B), and Tongil-type rice (C) ecotypes on grain-related traits. Black rectangles indicate the means of grain-related traits. GL: grain length, GW: grain width, GT: grain thickness, RLW: length to width ratio, TGW: 1,000-grain weight. Abbreviation of allele types are listed in Table 1. Means with same letters in a column are not significant at p<0.05 (ANOVA followed by DMRT).
자포니카 생태형에 포한된 흑미 품종은 Cj_b1, Cj_b2, Cj_b3 등 세 개의 대립유전자 조합이 존재하였다(Table 2). Cj_b1 (GW2-GS3_C-qGL3-qsw5_N-gs5-TGW6-GW7-GW8)은 Cj2, Cj_b2 (GW2-gs3-qGL3-qsw5_N-gs5-TGW6-gw7-GW8)는 Cj5와 동일한 구성의 조합이고 Cj_b3 (GW2-gs3-qGL3- qsw5_N-gs5-TGW6-GW7-GW8)은 Cj_b2에서 gw7이 GW7로 치환된 조합이다. 흑미 14품종 중 Cj_b1은 ‘흑광’ 1품종, Cj_b2는 ‘흑진주’ 등 7품종, Cj_b3는 ‘보석흑찰’ 등 6품종이 속해 Cj_b2와 Cj_b3 조합이 흑미의 대부분을 차지하였다. Cj_b2와 Cj_b3는 자포니카를 대표하는 Cj2와 Cj1에서 GS3_C가 gs3로 치환된 조합으로 현미 길이 등 입형 형질이 대부분 비슷하였고 너비를 제외하고 Cj_b1에 비해 형질 값이 큰 중립종에 중원형 입형 특성을 나타냈다(Fig 3B). 같은 자포니카 생태형인 흑미의 입형 특성을 구분하는 것은 gs3 대립유전자로 판단되며,유색미로 안토시아닌 등 색소의 착색에 따른 색택이 상품성의 중요한 역할을 하는 흑미에 있어서 종실이 길어지는 gs3형으로 편중된 품종이 개발된 것은 gs3로의 치환이 색택에 긍정적인 역할을 하여 육종 과정 중 gs3에 대한 선발압이 작용한 것이 아닌가 생각된다.
통일형 생태형은 GW2 유전자에서만 대립유전자의 다형성이 없었으며, 7개 유전자에서 13개 대립유전자 조합이 나타났다(Table 2). 자포니카 대립유전자 조합 Cj1과 동일한 CT1 (GW2-GS3_C-qGL3-qsw5_N-gs5-TGW6-gw7-GW8)을 제외한 CT2-CT13 등 12개 통일형 조합은 GW7-gw8 대립유전자형을 가지고 있어 자포니카 생태형과 다른 대립유전자 조합을 나타내며, CT1과 CT2 (GW2-GS3_C-qGL3- qsw5_N-gs5-TGW6-GW7-gw8)을 제외한 11개 통일형 조합은 GW7-gw8와 함께 qgl3 대립유전자형을 보유하고 있었다. CT1는 ‘녹양’과 ‘녹우’, CT2는 ‘목양’이 포함되어 있으며 다른 통일형 조합에 비해 현미 너비가 넓고 두께가 두꺼우면 무게가 무거웠다(Table 2, Fig. 3C). ‘녹양’, ‘녹우’, ‘목양’은 사료용 벼로서 교배조합에 국제 벼 연구소에서 도입된 열대 자포니카 계통이 포함되어 있어 입형 특성이 다른 통일형 품종에 비해 자포니카와 비슷한 것으로 생각된다(Ahn et al., 2018). CT3 (GW2-GS3_C-qgl3-qsw5_N-gs5- TGW6-GW7-gw8)은 33개 통일형 생태형 중 ‘한아름’, ‘큰섬’, 한아름2호’ 등 10개 품종(30.3%)이 포함되어 빈도가 가장 높으며, 자포니카 생태형 주요 대립유전자 조합으로 중단립종·단원형 입형 특성을 나타내는 Cj2에서 qGL3와 GW8이 qgl3와 gw8로 치환된 조합으로 평균 현미 길이, 너비, 두께, 천립중이 5.84 mm, 2.55 mm, 1.85 mm, 2.30, 20.3 g으로 중립종·중원형 입형 특성을 나타냈다. CT5 (GW2- GS3_B-qgl3-qsw5_N-gs5-TGW6-GW7-gw8)는 CT3에서 GS3_C가 GS3_B로 치환된 조합으로 ‘삼강’, 용문’, ‘남천’ 3품종이 포함되어 있으며 CT3과 현미 너비, 두께, 장폭비는 비슷하였으나 길이와 천립중이 감소하였고, CT4 (GW2-GS3_ B-qgl3-qsw5_N-GS5-TGW6-GW7-gw8)는 CT5에서 gs5가 GS5로 치환된 조합으로 ‘태백’, ‘용주’, ‘향미벼1호,’ ‘향미벼2호’ 4품종이 포함되어 있으며 CT5, CT3과 현미 너비와 두께는 비슷하나 길이가 길어 장폭비가 크고 천립중은 CT5보다 무겁고 CT3과 비슷한 수준이었다. CT6 (GW2- GS3_C-qgl3-qSW5-GS5-TGW6-GW7-gw8)과 CT7 (GW2-GS3_ C-qgl3-qsw5_I-GS5-TGW6-GW7-gw8)은 CT3에서 qsw5_N-gs5가 qSW5-GS5과 qsw5_I-GS5로 각각 치환된 조합으로 ‘다산’(CT6), ‘목우’와 ‘영우’(CT7) 품종이 포함되어 있으며, CT3에 비해 현미 길이와 너비가 다소 큰 편이었다. CT8 (GW2-GS3_B-qgl3-qsw5_I-GS5-TGW6-GW7-gw8)은 CT7에서 GS3_C가 GS3_B로 치환된 조합으로 ‘다산1호’와 ‘청우’가 포함되어 있고 CT7에 비해 현미 길이, 너비, 두께, 천립중이 다소 적은 편이였으며, CT9 (GW2-GS3_B-qgl3-qSW5- GS5-tgw6-GW7-gw8)은 국내육성 벼 품종 중 유일하게 tgw6를 가지고 있는 조합으로 ‘미면’ 1 품종만 여기에 속하였으며 통일형 주요 조합인 CT3에 비해 천립중 등 종실 크기에 관여하는 형질 값이 큰 편이었다. CT10 (GW2-GS3_ B-qgl3-qSW5-gs5-TGW6-GW7-gw8)은 CT5에서 qsw5_N이 qSW5로 치환된 조합으로 ‘한강찰’이 포함되어 있었고 CT5에 비해 천립중은 무거웠으나 다른 형질들은 비슷한 수준이었고, CT11 (GW2-GS3_C-qgl3-qSW5-gs5-TGW6-GW7-gw8)은 CT3에서 qsw5_N이 qSW5로 치환된 조합으로 ‘남풍’과 ‘팔방미’가 여기에 속하며 CT3에 비해 현미 길이가 길어지고 너비는 줄어들어 장폭비가 증가하고 천립중은 감소하였다. CT12 (GW2-gs3-qgl3-qsw5_N-gs5-TGW6-GW7-gw8)와 CT13 (GW2-gs3-qgl3-qSW5-GS5-TGW6-GW7-gw8)은 통일형 생태형 중 gs3를 보유한 조합으로 CT12는 CT3에서 GS3_C가 gs3로 치환된 조합으로 ‘안다’, ‘아름’, ‘세계진미’ 3품종이 속하였고 CT3에 비해 현미 너비 두께, 천립중은 비슷하고 길이가 길어 장폭비가 증가하였으며, CT13은 CT6에서 GS3_C가 gs3로 치환된 조합으로 ‘한강찰’이 여기에 속하며 현미 너비와 두께는 비슷하나 길이가 길어져 장폭비가 크고 천립중이 증가하였다.
종실의 크기와 형태는 여러 유전자가 관여하여 조절되는 양적형질로 약 400여개가 넘는 많은 양적형질유전자좌가 12개 염색체 전반에 걸쳐서 탐색되며, 유전자들의 상호작용인 상위성도 존재한다(Huang et al., 2013; Wang et al., 2015). 입형 관련 유전자가 형질에 미치는 영향을 정확하게 파악하기 위해서는 가급적 해당 유전자 이외에 다른 유전적 배경에 의한 간섭효과는 배제되어여 한다. 본 시험에서 GS3 대립유전자형 gs3, GS3_B, GS3_C이 입형 관련 형질에 미치는 영향 분석에서 현미 길이에 대해 전체 품종을 대상으로 할 경우에 gs3 > GS3_B > GS3_C형 순으로 기존의 연구결과와 일치하였다(Lee et al., 2015). 하지만 GS3 유전자 이외에 다른 유전자들은 대립유전자형이 같은 생태형별 대립유전자 조합간 비교에서 자포니카 Cj5 (gs3) > Cj1 (GS3_C), 흑미 Cj_b3 (gs3) > Cj_b1 (GS3_C), 통일형 CT13 (gs3) > CT6 (GS3_C)로 기능상실 대립유전자형인 gs3가 GS3_C형에 비해 길이가 길어지는 것은 기존과 같은 결과였으나, GS3_B와 GS3_C간 비교에서는 CT3 > CT5, CT7 > CT8, CT11 > CT10으로 GS3_C가 GS3_B형에 비해 길이가 길어 GS3_B가 GS3_C형 보다 길다는 기존과 전체 품종을 대상으로 한 결과와 달랐다. 이는 GS3_B 대립유전자를 보유한 품종은 12개로 적고 모두 통일형 생태형으로 편이되게 분포되어 전체 품종을 대상으로 분석할 경우에는 대다수를 차지하는 GS3_C형의 자포니카 품종의 입형 특성에 영향을 받아서 GS3_B형이 GS3_C형에 비해 길이가 긴 것으로 분석되었으나, 표본수가 비슷하고 같은 통일형 생태형의 대립유전자 조합에서의 비교에서는 GS3_C가 GS3_B형보다 길다는 다른 결과가 나타난 것으로 판단되어 이에 대한 검토가 필요할 것으로 생각된다.
자포니카 품종의 입형 관련 대립유전자 계보 분석
우리나라 자포니카 벼 품종들은 대부분 중단립종·단원형의 매우 협소한 입형 특성을 가지고 있으며, 협소한 유전적 배경으로 인하여 새롭게 개발되는 육성 계통이나 품종들의 입형은 중단립종·단원형 위주로 고착화되었다(Park et al., 2017). 협소한 입형 특성을 타파하기 위해서는 새로운 입형 관련 대립유전자의 도입이 필수적이며 이를 통해 다양한 입형 특성을 확보할 수 있다. 국내 육성 자포니카 품종의 대립유전자 조합은 7개로 qGL3, TGW6, GW8 유전자에 대한 다형성은 없었으며, 자포니카 대표 조합인 Cj1과 Cj2에 gw2, gs3, qSW5, GS5 대립유전자가 도입되어 다양성이 확대된 것으로 판단되어 이들 도입 대립유전자에 대한 계보 분석을 수행하였다. 종실이 대립화되는 gw2 대립유전자를 보유한 최초의 품종 ‘대립벼1호’(1993년 개발)의 교배조합은 Nihonmasari ms/BG 29로 ‘BG 29’ (Big Grain 29)는 천립중이 37.1 g인 대립형 일본 자원으로 ‘대립벼1호’의 gw2는 ‘BG 29’로부터 유래한 것으로 추정되며, 대립 쌀가루 품종인 ‘한가루’(교배조합 대립벼1호/설갱, 2016년 개발)의 gw2는 부본인 ‘설갱’이 GW2를 보유하고 있기 때문에 ‘대립벼1호’로부터 유래한 것으로 판단된다. 대립 찰벼인 ‘눈보라’(교배조합 익산433호/Miyadamamochi, 2006년 개발)의 gw2는 모본인 ‘익산433호’의 현미 천립중이 23.4 g으로 작아 일본 찰벼 ‘Miyadamamochi’에서 유래한 것으로 추정된다. 자포니카 품종 중 유일하게 qSW5 대립유전자를 보유고 있는 ‘아로미’(교배조합 새일미(밀양244호)/Princess Sari, 2016년 개발)는 ‘새일미’가 qsw5_N을 보유하고 있어 일본 향미 품종 ‘Princess Sari’ (교배조합 Kanto154/Kanto150)로부터 qSW5가 유래한 것으로 판단된다. ‘Princess Sari’의 qSW5 대립유전자는 모본인 Sari Queen (Kanto154; 교배조합 Nipponbare/Basmati370)가 qSW5를 보유하고 있는 것으로 알려진 Basmati370를 부본으로 이용하여 개발되어 이를 통해 도입된 것으로 생각된다(Ando et al., 2004; Lee et al., 2015). 종실 크기가 증가하였던 GS5 대립유전자를 보유하고 있는 자포니카 초다수 품종 ‘남일’(2002년 개발)은 교배조합이 일품/남양7호(교배조합 Wasetoramochi/EESD.7// 설악벼(철원21호))로 GS5는 일본찰벼 ‘Wasetoramochi’ 또는 유전자원 EESD.7로부터 유래한 것으로 추정되며, ‘새오대’(교배조합 그루벼/남일(수원472호), 2012년 개발)의 GS5는 ‘그루벼’가 gs5를 보유하고 있기 때문에 ‘남일’로부터 유래한 것으로 판단된다.
‘신동진’은 1999년 개발되어 2018년부터 우리나라에서 가장 넓은 면적에서 재배되고 있는 대표 품종으로 기존의 품종들에 비해 쌀알이 육안으로도 구분이 가능할 정도로 커서 차별화가 가능한 밥맛 좋은 우수한 품종이다. 쌀알 크기가 큰 ‘신동진’의 입형 특성은 다른 품종들과 비교되는 가장 큰 특징이나 아직까지 그 원인에 대해서는 밝혀진 바가 없다. 이번 연구 결과를 통해 ‘신동진’의 입형 특성은 종실 크기에 관여하는 GS3 유전자가 기존 대부분의 자포니카 품종들이 가지고 있던 GS3_C 대립유전자형에서 기능상실 대립유전자형인 gs3로 치환된 것에 기인함을 확인하였다(Table 2, Fig. 3). 기존 자포니카 품종이 보유하고 있지 않았던 gs3 대립유전자가 어떤 경로를 통해 ‘신동진’에 도입 되었는지를 추적하기 위해서 계보 분석을 수행하였다(Fig. 5). ‘신동진’은 ‘소비’(1999년 개발)와 동일 교배조합의 자매 품종으로 ‘화영’에 ‘YR13604Acp22’ 계통을 교배하여 육성되었다. ‘YR13604Acp22’는 ‘화영’에 고품질 쌀로 알려진 미국 품종 ‘Calrose’와 일본 품종 ‘Kinuhikari’를 교배한 F1을 삼원교배하여 약배양을 통해 육성된 계통으로 계보 분석을 위해 농업유전자원센터를 통해 분양 가능한 ‘Calrose’ 유전자원 8점(IT000534, IT000535, IT000536, IT000538, IT000539, IT000540, IT000541, IT000545)과 ‘Kinuhikari’ 유전자원 2점(IT173299, IT218283)을 확보하여 입형 관련 유전자형 분석을 수행하였다(Fig. 5A). GW2, GS3, qGL3, LGY3, qSW5, GS5, TGW6, GW7, GW8 등 9개 유전자에 대한 분자표지 검정 결과 GS3, qSW5, GW7에서 다형성이 발견되었다. ‘신동진’은 gs3-qsw5_N-gw7의 대립유전자형을 보유하고 있었고 ‘화영’은 GS3_C-qsw5_ N-gw7형을 나타냈으며 ‘Kinuhikari’ 유전자원은 GS3_C- qsw5-gw7 (IT173299)과 GS3_C-qsw5-GW7 (IT218283)형을 나타내 GS3_C를 가지고 있는 ‘화영’과 ‘Kinuhikari’는 ‘신동진’의 gs3 도입에 관여하지 않은 것으로 판단되었다. ‘Calrose’ 유전자원 8점의 대립유전자형은GS3_C-qsw5_N-gw7 (IT000534, IT000540), GS3_C-qsw5_N-GW7 (IT000535), GS3_C-qSW5-GW7 (IT000536, IT000538, IT000539, IT000545), gs3-qsw5-gw7 (IT000541) 등 4개였으며, 이 중 gs3 대립유전자를 가지고 있는 유전자원 IT000541이 탐색되었다. IT000541가 직접적으로 ‘YR13604Acp22’의 육성에 활용되었는지는 확인할 수 없고 IT000541 이외에 gs3를 보유한 다른 ‘Calorse’ 유전자원이 존재할 수 있으나, ‘신동진’의 gs3 대립유전자는 ‘화영’이나 ‘Kinuhikari’가 아닌 ‘Calrose’로부터 유래한 것으로 판단된다. ‘화영’과 ‘Kinuhikari’의 입형 특성은 자포니카 육성품종과 비슷한 중단립·단원형 특성을 나타냈으나, ‘Calrose’ (IT000541)와 ‘신동진’은 육안으로 구분이 가능할 정도로 종실 크기가 큰 입형 특성을 나타냈다(Fig. 5B). ‘신동진’과 ‘소비’가 1999년에 개발된 이후 이들 품종을 교배모본으로 이용하여 고품질 ‘평안’(2003년 개발), ‘다미’(2006년), 자포니카 초다수 ‘한마음’(2004년), ‘희망찬’(2011년), 찰벼 ‘백설찰’(2007년) 등 기존 자포니카 품종의 중단립종·단원형 입형과 구분이 가능한 장립종과 중원형에 근접하게 종실 크기가 증대된 중대립종 품종들이 개발되었다(Fig. 5C). ‘신동진’ 개발을 통한 국내 자포니카 품종으로의 gs3 대립유전자의 도입은 단순히 종실 크기의 증가뿐만 아니라 새로운 식감의 고품질, sink size 증대 통한 초다수, 가공적성 변경 특수미 등 국내 육성품종의 다양성을 확대하는데 큰 기여를 하였다.
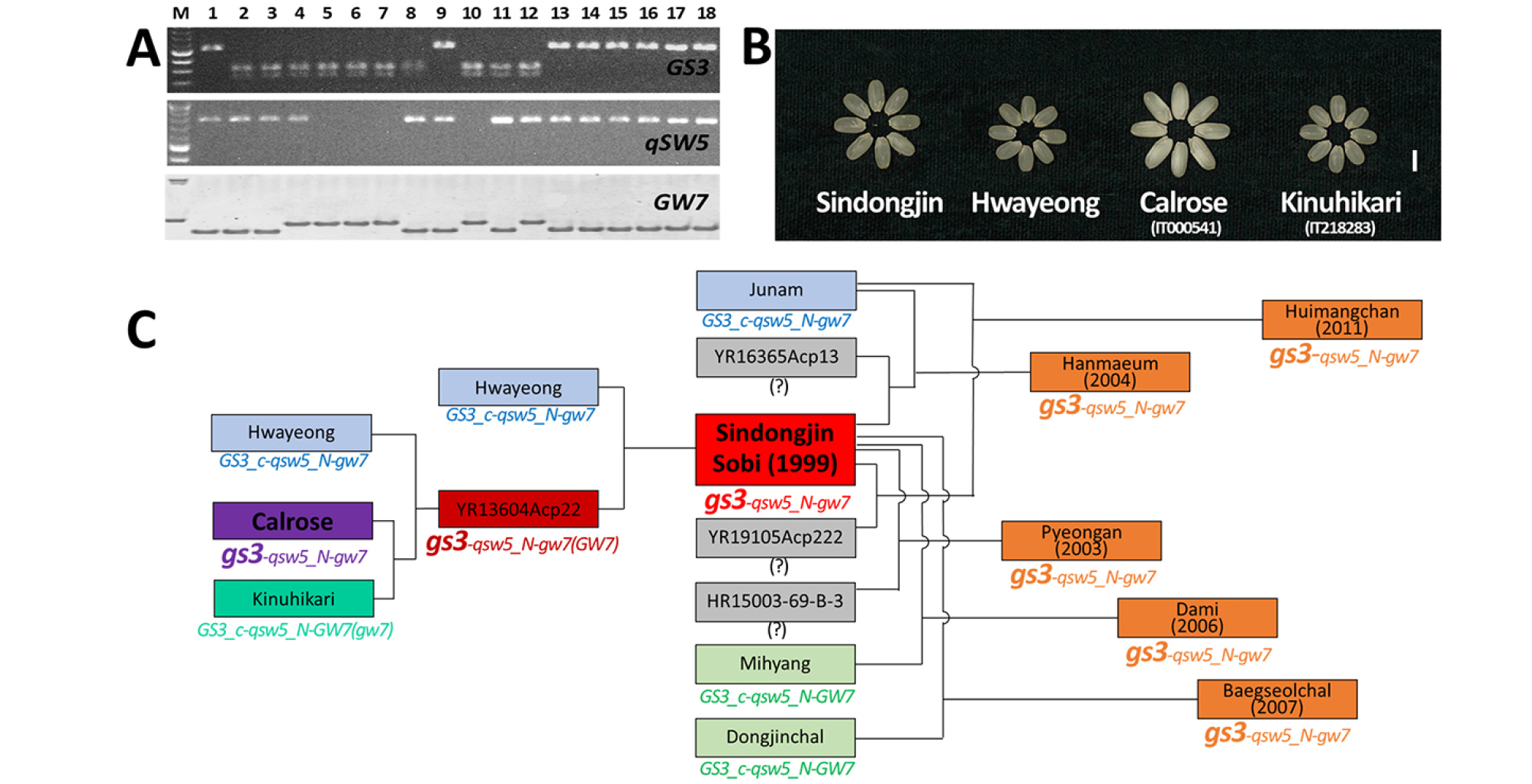
Fig. 5.
Deciphering the introgression of gs3 allele into Korean japonica rice varieties. PCR analysis to confirm the allele types of varieties involved in the pedigree of Sindongjin using gene specific DNA markers. (A). M: size marker, 1: Sindongjin, 2: Hwayeong, 3: Calrose (IT000534), 4: Calrose (IT000535), 5: Calrose (IT000536), 6: Calrose (IT000538), 7: Calrose (IT000539), 8: Calrose (IT000540), 9: Calrose (IT000541), 10: Calrose (IT000545), 11: Kinuhikari (IT173299), 12: Kinuhikari (IT218283), 13: Sobi, 14: Pyeongan, 15: Hanmaeum, 16: Dami, 17: Baegseolchal, 18: Huimangchan. Image of brown rice of Sindongjin and its ancestral parents (B). White bar indicates 5 mm. Allele flow of gs3 into Korean japonica rice varieties (C).
국내 육성 벼의 정체되어 있는 수량성 장벽과 기후변화에 따른 품질저하 극복을 위한 기반연구와 더불어 새로운 품질에 대한 소비자의 요구 충족과 쌀 산업 발전을 위한 사회적 변화를 견인하기 위해서는 우리나라 벼 품종의 입형 특성을 다양화해야 한다(Park et al., 2017). 이를 위해 대립형 국외자원을 활용한 자포니카 ‘입형 다양화 육종소재’의 개발 및 특성 분석, 수출쌀 전용품종 개발을 위한 자포니카 장립종·장원형 우량계통 ‘전주625호’ 개발 등 입형 특성을 다양화하기 위한 육종적 노력이 이루어지고 있다(Park et al., 2017; Park et al., 2018). 국내 육성 벼 품종에 대한 입형 관련 표현형과 유전자형 분석 결과는 우리나라 벼 품종의 입형 특성을 다양화하는데 기여할 것으로 기대된다.
적 요
국내 육성 벼 품종의 입형 특성은 협소한 유전적 배경을 가지고 있는 것으로 알려져 있다. 본 연구는 육성품종의 입형 관련 표현형과 유전자형을 분석하여 생태형에 따른 입형 특성과 대립유전자 효과를 파악하고, 자포니카 품종에 도입된 대립유전자의 기원을 확인하고자 수행되었다. 자포니카 225, 흑미 14, 통일형 생태형 33품종 등 272품종에 대해서 현미 길이, 너비, 두께, 장폭비, 천립중의 표현형과 GW2, GS3, qGL3, qSW5, GS5, TGW6, GW7, GW8 등 8개 입형 관련 유전자형을 분석하였다. 자포니카 품종은 중단립종에 단원형, 흑미와 통일형 품종은 중립종에 중원형 입형 특성을 나타냈다. 표현형에 대한 군집분석을 통해 자포니카 품종 대부분으로 구성된 그룹 1, 흑미와 통일형 품종 위주로 구성된 그룹 2, 자포니카 품종이 포함된 그룹 3 등 세 그룹으로 나눌 수 있었다. 그룹 1은 현미 너비와 두께, 그룹 2는 장폭비와 길이, 그룹 3은 천립중에 의해 영향을 많이 받아 구분되며 그룹 1은 중단립종·단원형, 그룹 2는 중립종·중원형, 그룹 3은 장립종·단원형 입형 특성을 나타냈다. 입형 관련 대립유전자형 분석 결과 gw2 (빈도수 1.1%)와 tgw6 (0.4%) 대립유전자는 매우 드물었으며, qgl3와 gw8는 통일형 생태형에서만 존재하였고 자포니카 품종의 qSW5 유전자형은 qsw5_N이 대부분을 차지하였다. 생태형별 대립유전자 조합의 수는 자포니카 7개(Cj1-Cj7), 흑미 3개(Cj_b1-Cj_b3), 통일형 13개(CT1-CT13)로 자포니카에 비해 품종수가 적은 통일형 생태형이 더 다양하였다. 자포니카 품종의 대표 대립유전자 조합은 자포니카 Cj1, 2 (GW2- GS3_C-qGL3-qsw5_N-gs5-TGW6-gw7(GW7)-GW8)로 여기에 gw2, gs3, qSW5, GS5 대립유전자가 도입됨으로써 다양성이 확대되었다. 흑미 품종의 대표 대립유전자 조합은 Cj_b2, 3 (GW2-gs3-qGL3-qsw5_N-gs5-TGW6-gw7(GW7)-GW8)로 자포니카 대표 조합에서 GS3_C가 gs3로 치환된 조합이다. 통일형 생태형은 GW2 유전자만 다형성이 없었고 7개 유전자에서 13개 대립유전자 조합이 확인되었으며 대표조합은 CT3 (GW2-GS3_C-qgl3-qsw5_N-gs5-TGW6-GW7-gw8)이다. 우리나라 대표 품종인 ‘신동진’의 입형 특성은 자포니카 대립유전자 조합 Cj2에서 gs3가 도입됨으로써 중대립화되었고, gs3는 미국품종 Calrose로부터 유래한 것으로 판단된다. 국내 육성 벼 품종에 대한 입형 관련 표현형과 유전자형 분석 결과는 우리나라 벼 품종의 입형 특성을 다양화하는데 기여할 것으로 기대된다.




