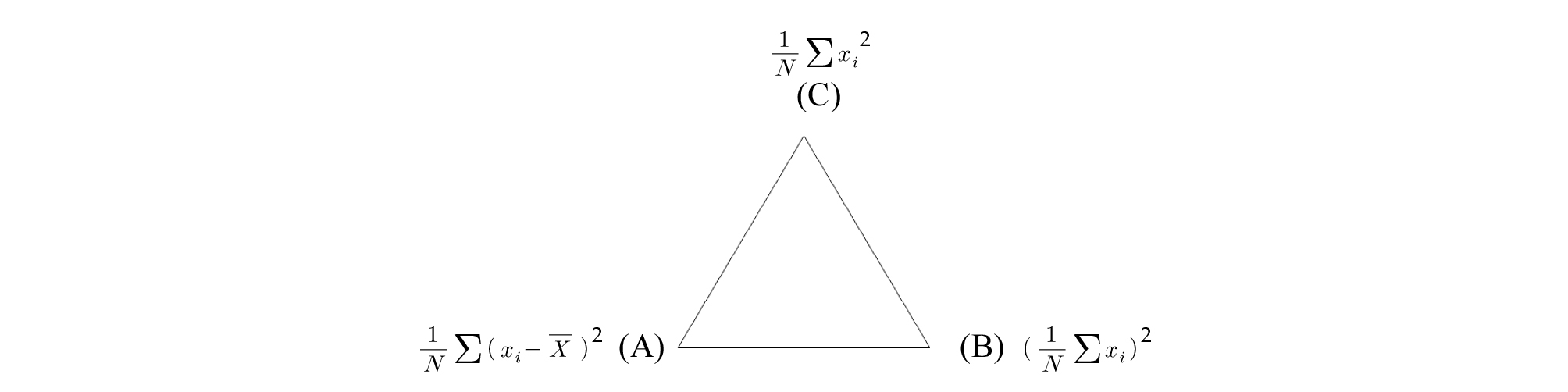서 론
재료 및 방법
시험자원 선정 및 처리
NIRS 검량식 작성 및 검증
NIRS를 이용한 아밀로스 및 단백질 함량 측정
자원들의 다양성 지수 계산
확률밀도함수와 정규분포
분산분석(ANOVA)
Duncan’s multiple range test (DMRT)
결과 및 고찰
NIRS 검량식 작성 및 검증
벼 유전자원의 집단분석
벼 유전자원의 국가별 품종 기준(ANOVA)
벼 유전자원의 국가별 특성(DMRT)
적 요
서 론
쌀(Oryza sativa L.)은 세계 3대 곡물중의 하나이며 우리나라를 포함한 아시아 지역에서 전 세계 쌀 생산량의 90% 이상이 생산되는 중요한 식량원이다(Juliano, 2003; Kim et al., 2012). 쌀의 주요성분은 아밀로스와 단백질로 구성되며 품종에 따라 성분 함량이 다르다. 쌀을 주식으로 하는 나라에서는 이들 성분의 특정 함량을 가진 쌀 품종을 선호하는 경향이 있으며, 따라서 품종육성연구의 경우 아밀로스 성분이 중요 지표 자료로 활용되기도 한다(Kim, 2004). 아밀로스와 단백질은 쌀의 미질을 결정하는 중요한 이화학적 특성이고 품질관련변수이며 취반특성 뿐만 아니라 물리적 특성에도 영향을 준다(Son et al., 2002; Champagne et al., 1997).
농산품의 경우 주요구성 성분인 지방(C-H), 수분(O-H), 단백질(N-H, S-H)이 근적외선을 흡수하므로 근적외선 분광분석법을 이용해 이들 성분의 동시 분석이 가능하다(Williams et al., 1987). 시료 내 다양한 성분들의 근적외선 흡수파장대가 서로 중첩되는 현상 또는 시료 입자 크기, 밀도 등과 같은 물리적 요인들에 의해 스펙트럼의 변화가 일어난다. 이러한 변화에 의한 분석 오차를 줄이고 상호 중첩된 파장들을 분리하기 위해 NIRS전용 프로그램을 사용하여 스펙트럼을 보정하는 등의 수학적 전처리 과정을 수행하게 된다(Ahn & Kim, 2012). 이후 습식분석결과와 근적외선 스펙트럼간의 상관관계를 회귀분석을 통해 분석하여 검량식 작성 등을 통해 최종적으로 NIRS 예측모델을 개발한다. 개발된 모델이 적용된 NIRS를 이용하여 유전자원의 성분 함량을 측정하고 얻어진 data는 통계처리 과정을 거쳐 함량 특성 분석에 활용된다. 농업분야에서 많이 사용되는 통계처리방법에는 분산분석(ANOVA)과 Duncan’s다중검정(DMRT)이 있다. 분산분석은 세 개 이상 집단들 간의 평균 차이를 비교하기 위하여 집단 간 분산과 집단 내 분산을 계산하고 이들 상호간의 비를 이용한다. 집단의 표본평균들이 동일한 모집단에서 나온 것인지를 검정하며, 실험에 관련된 요인들 가운데 가장 큰 영향을 끼치는 것을 찾아내는 분석법이다(Kim, 2016; Pak & Oh, 2010; Park et al., 2016). ANOVA를 통해 집단들 간의 평균들이 유의미하게 차이가 있다는 결과를 얻게 되면 이후 DMRT를 사용하여 각 집단들 간의 평균차이를 상호 비교하여 구체적으로 어느 집단 간에 차이가 있는지를 검정한다. 이러한 통계처리는 연구 결과의 신뢰도를 높일 수 있으므로 다양한 연구 분야에 적용되고 있다.
NIRS를 이용한 재래종 자원의 성분분석 결과를 토대로 각 성분 구간별 확률값을 구하여 막대그래프로 표시한 후 이를 바탕으로 x축은 연속확률변수로 y축은 확률값을 계급값으로 나눈 수치로 나타내어 확률밀도함수의 면적값이 확률값이 되도록 표시하였다. 이를 근거로 하여 정규분포 함수에 적용하여 실측치와 이상치의 차이를 확인하였다. 이에 대한 보고는 선행 연구논문에 언급하였다(Oh et al., 2019). 유전자원은 근본적으로 자원에 대한 다양성을 확보하는데 주력을 하고있으며, 일반에게 직접적으로 관련되는 생산성, 저항성 등도 중요하지만 다양한 특성을 나타내는 것이 유전자원으로써 가치를 부여하기 때문에 Variability Index Value (VIV)를 집단별로 분석하여 그 정도를 표시하였다. 대부분의 논문들에서 통계프로그램을 사용하여 계산된 최종결과만을 간략하게 언급하거나 단순한 통계처리결과만을 나타내는데 그치고 있으며, 적용된 통계처리방법에 관한 상세한 설명을 생략하여 정확한 이해가 어려운 실정이다. 따라서 통계처리방법에 관한 이해를 돕고 효율적인 적용을 통해 연구결과의 신뢰성을 높이고 객관성을 확보하고자 기초이론을 해석함과 동시에 농업유전자원센터에 보존되어있는 재래종 벼 유전자원의 아밀로스 및 단백질 함량에 관한 NIRS 성분 분석 자료를 바탕으로 Normal Distribution, VIV, ANOVA, DMRT를 수행하였다. 보존자원의 다양성과 성분함량에 따라 자원 집단을 구분하고, 국가별 재래종 자원들에 대하여 통계분석을 하였으며, NIRS 대량평가체계를 기반으로 자원들 간의 집단분석 결과를 기초자료로 제시하고자 이 연구를 수행하였다.
재료 및 방법
시험자원 선정 및 처리
본 실험에 사용한 국내외 재래종 벼 유전자원은 농업유전자원센터 GMS 프로그램을 이용하여 한·중·일 등 41개국에서 수집된 자원을 대상으로 IT Number 기준으로 정렬하여 4,948자원을 선발하였고, 선발자원 각각의 종자 50립을 실험실용 현미기로 탈영시킨 후 사미, 피해립을 제거하여 현미립을 준비하였다. 이후 분쇄기(Bistro electric coffee grinder, BodumⓇ)를 사용하여 26,000 rpm 조건에서 약 2분간 분쇄한 후 300 μm sieve를 통과하는 균일한 현미가루 상태로 만든 후 NIRS를 이용하여 아밀로스함량과 단백질함량을 측정하였다. 벼 유전자원에 대한 고유특성을 최대한 반영하기 위하여 현미를 대상으로 분석을 실시하였다. 선발자원 중 아밀로스 함량 10% 이상의 메벼 4,380자원만을 택하여 아밀로스 및 단백질 함량분포 분석과 통계분석을 실시하였다(Table 1). 수분함량 분석은 곡류 및 곡류제품-수분함량측정(산업통상자원부 국가기술표준원, 표준번호: KS H ISO 712)을 참조하여 수분측정기(MOC63u, Shimadzu, Japan)를 사용하여 130°C 온도조건에서 측정하였다. 현미시료들은 13°C 저온저장고에 보관하였으며 수분함량이 14% 이하로 유지됨을 수분측정기로 분석하여 확인하였다.
Table 1. Distribution of landrace rice germplasm based on geographical origin.
NIRS 검량식 작성 및 검증
벼 유전자원의 아밀로스 및 단백질 성분 분석용 NIRS 예측모델을 선행연구에서 개발하고 미지시료 적용가능성을 검증하였다(Oh et al., 2017a). 현미가루 상태의 벼 511자원에 대한 근적외선 스펙트럼은 실온조건하의 가시광선 및 근적외선 대역(400-2500 nm)에서 한 자원 당 3반복 scanning하여 얻었다.
이후 검량식 작성을 위하여 벼 자원 별 아밀로스함량 및 조단백질 함량을 입력하여 습식분석값과 근적외선 스펙트럼을 파일형태로 저장하였다. WINISI Ⅲ project manager의 global equation 기능을 사용하여 습식분석값과 NIRS 스펙트럼 간의 통계적 해석과 상관관계분석을 통해 검량식을 작성하였다. 작성된 검량식 중 습식분석값을 가장 잘 예측할 수 있는 최적 NIRS 예측모델은 RSQ (R2), Standard error of calibration (SEC), Standard error of prediction (SEP), Slope, Bias, Standard error of cross-validation (SECV) 및 One minus the ratio of unexplained variance to total variance (1-VR) 등의 통계치를 고려하여 선별하였다. 최적 NIRS 예측모델의 미지시료 적용가능성을 검증하기 위해 검량식 작성에 이용되지 않은 자원들을 대상으로 근적외선 스펙트럼 측정과 아밀로스 및 단백질 함량 분석을 수행하여 validation file을 작성하였다. 작성된 validation file은 WINISI Ⅲ project manager (FOSS, ver. 1.50e)의 monitor result 기능을 사용하여 Slope, RSQ, SEP, Bias 등의 통계치를 계산하였고 이를 바탕으로 NIRS 예측모델의 미지시료 적용가능성과 정확성을 검증하였다.
NIRS를 이용한 아밀로스 및 단백질 함량 측정
현미가루시료 약 600 mg을 micro insert ring을 끼운 mini sample cup에 채운 후 sample cup backs를 이용하여 시료 내 공극을 없애준 후 실온조건 가시광선 및 근적외선 대역(400-2500 nm)에서 한 자원 당 2반복하여 스펙트럼을 측정하였다. 스펙트럼 측정은 NIRS (FOSS, XRD near-infrared)를 사용하였고, 아밀로스 및 단백질 함량 계산은 개발된 NIRS 예측모델이 입력된 NIRS 구동 전용프로그램인 ISI scan (FOSS, ver. 4.2.0)을 사용하였다.
자원들의 다양성 지수 계산
다양성 지수는 자원들 간의 차이를 나타내기 위한 수단이지만, 수치화된 지표가 없으므로 분자생물학분야에서 사용되는 계산식을 적용하여 이를 다양성지수로 표시하고자 하였다. 아밀로스와 단백질 함량구간을 2% 함량단위로 나누고 각 함량구간에 속하는 자원수(ni)를 전체 자원수(4,380)로 나누어 다양성 확률값()을 계산했다. 다양성 확률값 각각을 제곱하고 모두 합산 후 1에서 뺀 값으로 다양성지수 VIV (Variability Index Value)를 구하였다. 집단 내 개체들의 특성을 표현하는 유전자는 2n 형태의 대립유전자로 존재한다. 유전적 다양성 계산에서는 대립유전자를 homogeneous type으로 가정하기 때문에 값은 homogeneous type의 유전자가 발현될 확률을 의미하며, 이를 1에서 뺀 값은 heterogeneous type의 유전자가 발현될 확률이다. 따라서 다양성 지수가 1에 가까울수록 heterogeneous type의 유전자가 발현될 확률이 크다는 의미이므로 집단의 다양성은 커진다.
Variability Index Value =
확률밀도함수와 정규분포
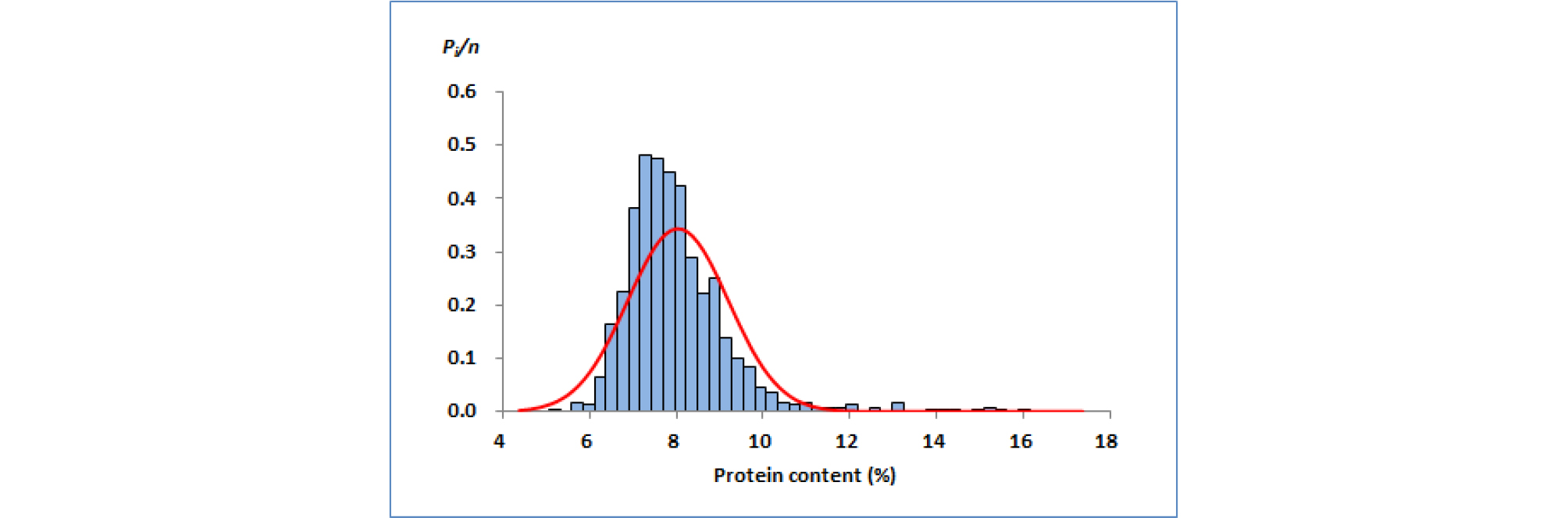
재래종 메벼 4,380(N) 자원에 대하여 아밀로스 및 단백질 함량 분포를 나타내는 도수분포표와 확률밀도를 나타내는 히스토그램을 구하였다. 계급폭(n, class width)은 아밀로스는 1, 단백질은 0.25, 계급은 아밀로스는 1 함량구간, 단백질은 0.25 함량구간으로 하였다. 이를 바탕으로 엑셀프로그램(Excel 2010)을 이용하여 히스토그램을 그렸다. 아밀로스 및 단백질 함량의 확률밀도함수를 나타내기 위해 히스토그램의 y축 값은 확률밀도를 나타내는 pi (확률)/n (계급폭)값으로, x축 값은 계급값들을 연속확률변수형태(contents range)로 표시하였다. 단백질 함량 도수분포표(Table 2)로부터 엑셀프로그램을 이용하여 확률밀도를 나타내는 히스토그램을 그렸다(Fig. 1). 각 단백질 함량구간의 에 계급폭 값 0.25를 곱하여 모두 합하면 각 함량구간의 pi (확률값)의 총 합과 동일한 값인 1이 된다. 자원 집단의 확률밀도함수가 정규분포와 유사할 경우 정규분포의 표준화 과정을 통해 특정 함량구간의 확률값을 구할 수 있고 전체 자원수를 확률값에 곱하면 특정 구간의 자원 비율을 계산할 수 있다. NIRS 측정결과 얻어진 함량 자료를 도수분포표로 정리하여 이를 바탕으로 계단모양의 히스토그램을 곡선 형태의 확률밀도함수로 표현되도록 하여 부드러운 곡선 형태를 이루는 정규분포와 비교하여 함량별 자원분포 특성을 확인하였다. 정규분포곡선은 재래종 전체 자원에 대한 아밀로스 및 단백질 함량의 평균값과 분산값을 구한 후 엑셀프로그램의 정규분포함수(NORM.DIST)에 이들 값들을 입력하여 구하였다. 정규분포는 하나의 이상적인 히스토그램이고 가장 중요한 연속확률분포이며 대부분의 통계이론의 기초를 이룬다. 실제 실험에서 관찰되는 많은 확률값들이 정규분포를 따르는지 확인하기 위하여 정규분포 함수식에 적용하여 비교하였다.
Table 2. Frequency distribution table of protein contents of landrace rice germplasm.
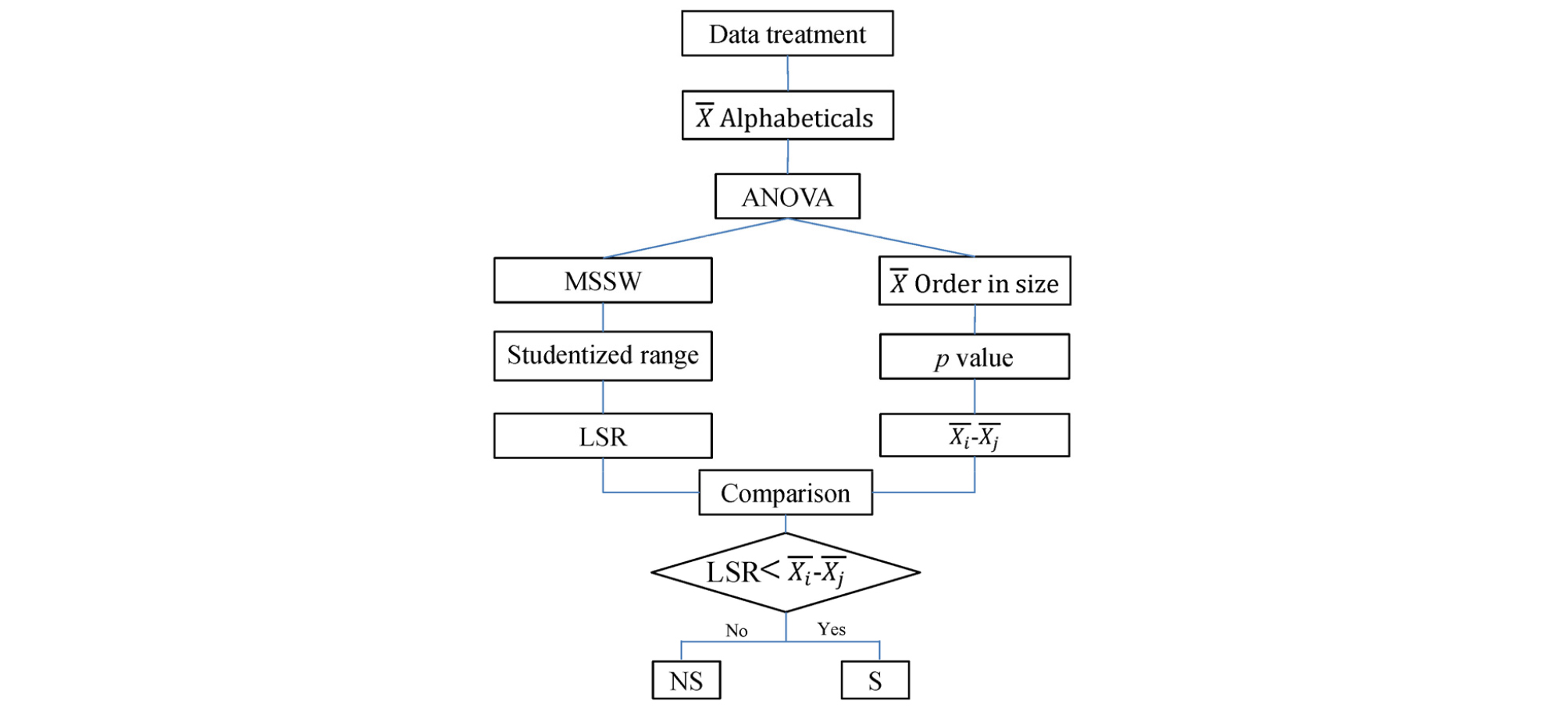
분산분석(ANOVA)
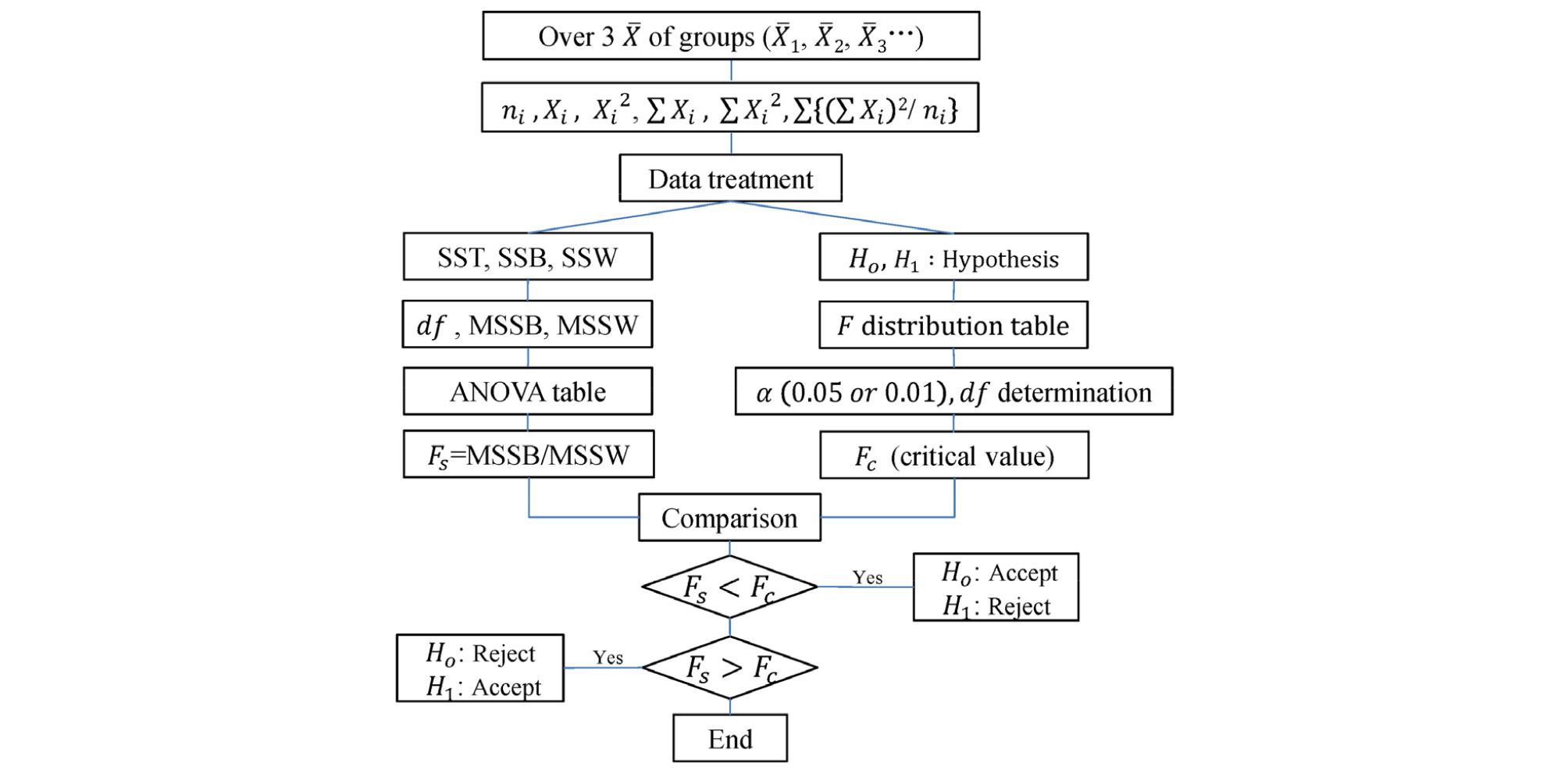
재래종 메벼 4,380자원 중 높은 자원비율을 나타낸 한국 원산지 1,032자원, 북한 원산지 994자원, 일본 원산지 800자원, 중국 원산지 528자원을 대상으로 분산분석을 이용하여 평균함량들 간의 유의성을 평가하였다. 독립변수는 국가별 자원 집단(t)으로, 종속변수는 아밀로스와 단백질 함량(X)으로 설정하였다. 세 집단(그룹) 이상의 집단 간 평균을 비교할 경우 분산분석(analysis of variance, ANOVA, F-검정)을 실시하게 되는데 F-분포를 이용하여 집단 간의 분산과 집단 내의 분산을 비교하여 평균들 간 차이유무를 검정하는 방법이다. ANOVA 검정 절차를 아래 Fig. 2에 나타냈다. 편차를 기반으로 하는 분산은 평균을 중심으로 관찰값 들이 얼마나 넓게 분포하는지를 나타내는 통계량으로 계산과정에서 모든 관찰값이 사용되기 때문에 평균과 함께 가장 많이 쓰인다. xi는 관찰값, N은 관찰값 수, 는 관찰값 평균이다. 편차는 표본평균()과 관찰값(xi)과의 차이를 말하며 분산은 편차제곱의 평균으로 정의되며, 관찰값 제곱의 평균에서 평균의 제곱 값을 뺀 값이다. 편차제곱합은 각 관찰값에서 평균을 뺀 편차를 제곱하여 모두 합한 값이다. 편차제곱합은 관찰값들 간에 차이가 클수록 큰 값이 나온다. 따라서 편차제곱합을 이용하는 분산은 관찰값의 분포정도를 나타내는 통계량으로서 그 표현력이 크다고 말할 수 있다. Fig. 3에서 분산(편차제곱의 평균), 평균의 제곱, 제곱의 평균 간 상호관계와 관계식을 나타냈다.
분산분석은 귀무가설 설정, 제곱합과 평균제곱합 계산, 분산분석표 작성, 유의수준과 기각값 설정, 귀무가설 채택 또는 기각 여부 결정, 분산분석 결과 해석 과정을 거친다. 귀무가설(H0)은 ‘모든 평균은 다 같다’이고, 대립가설(H1)은 ‘평균들이 모두 같지는 않다’이다. 가설검정과정에서 H0이 맞지만 기각하게 되는 제Ⅰ종 오류가 발생할 확률의 최대 허용한계를 유의수준(α)이라고 한다. α=0.01은 제Ⅰ종 오류의 최대 크기가 0.01 즉, 잘못된 판단을 할 확률이 0.01라는 것이며 ‘1%유의수준’이라고 말한다. ‘각각의 관찰값(xi)은 전체평균(), 군간 편차(), 군내 편차()의 합과 같다’는 식 (1)의 정의는 분산분석의 이론적 배경이다. 식 (1)을 식 (2)와 같이 정리한 후 양변을 제곱하고 전체 관찰값을 대상으로 계산하면(∑∑를 적용하면), 관찰값과 평균의 차이인 편차 총합은 0이 되므로 식 (3)을 얻을 수 있다. 를 총제곱합(SST)으로, 를 군간제곱합(SSB)으로, 를 군내제곱합(SSW)으로 정의되므로 SST=SSB+SSW 관계가 성립한다(식 4). 제곱합 들 간의 상호관계와 일반개념을 그림으로 나타냈다(Fig. 4). X는 확률변수(아밀로스 또는 단백질 함량)를, ∑xi는 한 집단 내 관찰값 들의 총합을, ∑∑xi는 전체 집단 내 관찰값 들의 총합을 의미하며 ∑x(=∑xt)과 동일한 표현이다. 는 전체 관찰값의 평균을 나타내며 는 한 집단 내 관찰값 들의 평균을 나타낸다. n은 전체 집단(그룹)의 수, ni는 한 집단 내 관찰값의 총 개수(집단의 크기)를 N은 전체 집단 내 관찰값의 총 개수를 의미하며 n과 ni의 곱의 값과 동일하다. SSB의 정의식은 식 (5), SSW의 정의식은 식 (6)와 같이 유도된다. 각 제곱합들의 정의식(좌변)과 실제 계산에 사용되는 계산식(우변)을 식 (7), 식 (8), 식 (9)에 나타냈다. 실제 분산분석을 위한 각 제곱합들의 계산은 항이 SST와 SSB에 동일하게 나타나서 계산 과정이 단순해지므로 SST와 SSB를 먼저 구한 후 SST=SSB+SSW 관계를 활용하여 SSW를 구한다.
| $$x_i=\overline{X_t}+(\overline{X_i}-\overline{X_t})+(x_i-\overline{X_i}) | 식 (1) |
| $$x-\overline{X_t}=(\overline{X_i}-\overline{X_t})+(x_i-\overline{X_i})$$ | 식 (2) |
상기 식 (2)의 양변을 제곱하여 전개하면,
{는 상수}
위 식의 좌변과 우변은 아래와 같이 SST, SSB, SSW로 정의된다.
(∵nin=N, n:집단(그룹)의 수, ni:한 집단 내 관찰값 수, N:전체 집단 내 관찰값 수)
위 식들을 요약하면 아래 식 (7), (8), (9)와 같다.
제곱합(SST, SSB, SSW)은 각각의 자유도를 가진다. 전체 집단의 수를 K (∑Group), 전체 집단의 크기(전체 집단의 확률변수 총 개수)를 N이라고 하면 SST 자유도는 N−1, SSB는 K−1, SSW는 N−K이다. SST 자유도는 SSB 자유도와 SSW 자유도의 합이다. 편차제곱합을 자유도로 나눈 것을 평균편차제곱합(MSS)이라 하며 MSSB와 MSSB로 분류한다. 군간평균제곱합은 , 군내평균제곱합은 이다. 검정통계량(Fs)은 군간평균제곱합을 군내평균제곱합으로 나눈 값이며 아래 식과 같다.
위 과정에서 계산된 통계량들을 정리한 분산분석표는 Table 3과 같다.
Table 3. ANOVA formula definition.
| SV | df | SS | MSS | Fs |
| SSB | K−1 | |||
| SSW | N−K or K(n−1) | |||
| SST | N−1 |
Duncan’s multiple range test (DMRT)
분산분석과정에서 귀무가설을 기각되어 각 집단 간 ‘평균들이 모두 같지는 않다’는 가설이 성립되면 이는 두 개 이상의 평균이 같지 않다는 뜻이며 어느 집단 간의 평균들이 차이가 있는지는 알 수 없다. 이 같은 경우 평균의 다중비교 방법을 사용하여 각 집단들 간 평균 비교를 할 수 있다. 이론적 배경은 여러 개의 평균들을 상호 짝을 지어 반복 비교하더라도 제1종 오류(평균간 차이가 없음에도 평균간 유의차가 있는 것으로 판단)가 일정 유의수준(0.01 또는 0.05)을 넘지 않는 구간(유의범위)을 정하여 평균간 차이와 비교하는 방법이다. 평균 간 차이가 유의하다고 판단할 수 있는 ‘최소유의차(LSD)’ 또는 ‘최소유의범위(LSR)’를 구하여 두 평균 간 차이를 비교하여 두 평균간 차이가 최소유의차 또는 최소유의범위보다 크면 두 평균은 유의하게 다른 것이고 작으면 유의차가 없는 것이다. Least Significant Difference (LSD)는 t 검정이며 이는 두 집단 간 비교에 이용된다. 따라서 다중검정에 적용할 경우 제1종 오류를 범할 위험성이 있다. 이를 보완한 방법이 DMRT이며 비교하는 평균들 간의 거리에 따라 서로 다른 유의범위를 적용한다. 농학 연구 분야에서는 주로 LSD와 DMRT가 많이 적용되며 본 연구에서는 DMRT를 사용하였다. DMRT는 평균을 크기순으로 정렬하고 가장 큰 평균부터 시작하여 가장 작은 평균까지 순차적으로 비교하게 되는데 이때 기준이 되는 값이 필요하다. 이 값을 Least Significant Range (LSR)이라고 하며 전체 평균수(전체 집단수, K) 보다 1개 적은 LSR값이 필요하다. LSR은 아래 식 (10)과 같이 정의된다. 는 표준오차이며 군내평균제곱합(MSSW)을 집단의 크기(n)로 나눈 수치의 양의 제곱근 값이다(식 (11)). 각 집단의 크기(n)가 서로 다를 경우는 식 (11)의 n대신 nh값을 적용한다. Significant Studentized Range (SSR)값은 오차자유도, 전체 평균수, 유의수준(α)을 정한 후 ‘다중검정을 위한 스튜던트화한 범위’표를 참조하여 구할 수 있다. 예를 들면 오차자유도 60, 유의수준(α) 0.05, 전체 집단수 4인 경우 SSR값은 2.83, 2.98, 3.08이다.
| $$LSR=SSR\times S_\overline x$$ | 식 (10) |
| $$S_\overline x=\sqrt{\frac{MSSW}{n(or\;n_h)}}\;,\;n_h=\frac K{\sum{\displaystyle\frac1{n_i}}}$$ | 식 (11) |
※ 전체 집단수: 3, 집단의 크기: 438, 308, 131이면
식 (10)에서 구한 LSR값을 크기순으로 나열하고 각 집단의 평균값을 크기순으로 나열하는 것으로 유의성검정과정을 시작한다. 가장 큰 평균에서 가장 큰 LSR값을 뺀 값은 가장 큰 평균과 나머지 평균들을 비교하는 기준값이고 이 보다 작은 평균값들이 존재하면 가장 큰 평균값과 5% 유의수준에서 유의차가 있다고 판단하고 가장 큰 평균값 아래 밑선을 긋거나 위 첨자 영문 소문자로 표시한다. 두 번째 큰 평균에서 두 번째 큰 LSR값을 뺀 값은 두 번째 큰 평균과 나머지 평균들을 비교하는 기준값이고 이 보다 작은 평균값들이 존재하면 두 번째 큰 평균값과 5% 유의수준에서 유의차가 있다고 판단하고 두 번째 큰 평균값 아래 밑선을 긋거나 위 첨자 영문 소문자로 표시한다. 이와 같은 과정을 모든 LSR값을 사용하여 검정과정을 끝낼 때 까지 반복한다. 만일 비교 기준값 보다 큰 평균값들이 존재하여 평균간 차이가 유의하지 않는 경우에는 비교하는 평균값들 아래 밑선 또는 동일한 문자표시를 하여 유의하지 않는 평균값을 가지는 동일 집단임을 표시한다.
DMRT에 의한 검정절차를 Fig. 5에 요약했다. 룹별 관찰값 정리와 평균값계산, 분산분석표 작성을 통해 집단 간 유의성이 확인되면(1단계) DMRT를 통해 각 집단 간 평균을 비교할 수 있다. 집단들의 평균값을 내림차순으로 정렬한 후 집단들이 이루는 평균값 구간에 존재하는 평균값 개수(집단개수, p 값)를 확인하여 DMRT에 필요한 p값을 정한다(2단계, Table 4). 평균 간 비교를 위한 기준이 되는 LSR값은 상기 식 (10), 식 (11)에 의해 구할 수 있다. SSR값은 p값, 검정유의수준(α), 오차자유도(SSW의 df)를 결정하면 ‘다중검정을 위한 스튜던트화한 범위’표를 참조하여 구할 수 있다. 는 표준오차이며 분산분석표의 MSSW값과 집단의 크기를 알면 식 (11)에 의해 구할 수 있다(3단계, Table 5). 각 집단들의 평균값()과 LSR값들을 크기순으로 나열하여 DMRT 표를 작성한다(4단계, Table 6). 평균()과 LSR값의 차(│−LSR0.01│)를 기준값으로 사용하여 다른 집단의 평균과 비교하거나 집단 간 평균 차(││)와 LSR0.01 값을 비교하는 두 가지 방식을 사용할 수 있다. 평균과 LSR값의 차(│−LSR0.01│)를 기준값으로 하는 경우 기준값이 다른 집단의 평균보다 크면 유의성이 있다고 판단하고 작으면 유의성이 없다고 판단한다. 집단 간 평균 차(││)와 LSR0.01값을 비교하는 경우 집단 간 평균간 차보다 LSR값이 크면 유의성이 없다고 판단하고 작으면 유의성이 있다고 판단한다. DMRT 결과를 실험 목적에 맞게 해석 한다(5단계, Table 7).
Table 4. Descending arrangement of average values () in each group and the determination of Studentized range p-value.
|
Alphabe- ticals | Array ⇨ |
Order in size | p value ⇨ |
Reference Statistics | B | C | E | A | D | ||
| A | 13.29 | B | 30.86 | D | 5 | 4 | 3 | 2 | |||
| A | 4 | 3 | 2 | ||||||||
| B | 30.86 | C | 29.14 | ||||||||
| E | 3 | 2 | 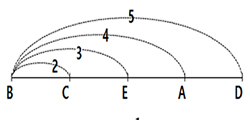 <p value> | ||||||||
| C | 29.14 | E | 21.00 | ||||||||
| C | 2 | ||||||||||
| D | 11.00 | A | 13.29 | ||||||||
| B | |||||||||||
| E | 21.00 | D | 11.00 | ||||||||
Table 5. Calculation of LSR value (at significance level α=0.01).
| pz | 2 | 3 | 4 | 5 |
| SSR (df=30, α=0.01) | 3.89 | 4.06 | 4.16 | 4.22 |
| LSR0.01(SSR×) | 3.89×1.10y=4.28 | 4.06×1.10=4.47 | 4.16×1.10=4.58 | 4.22×1.10=4.64 |
yMean .
Table 6. Creating DMRT table by comparing with and |−LSR0.01| value in each group.
Table 7. Interpretation of DMRT result.
| DMRT Result | Interpretation |
| B CEA D |
Equal to B and C at level of 1% Equal to A and D at level of 1% Grouping as A&D, B&C, and E at level of 1% |
| Aa Bb Cb Da Ec |
결과 및 고찰
NIRS 검량식 작성 및 검증
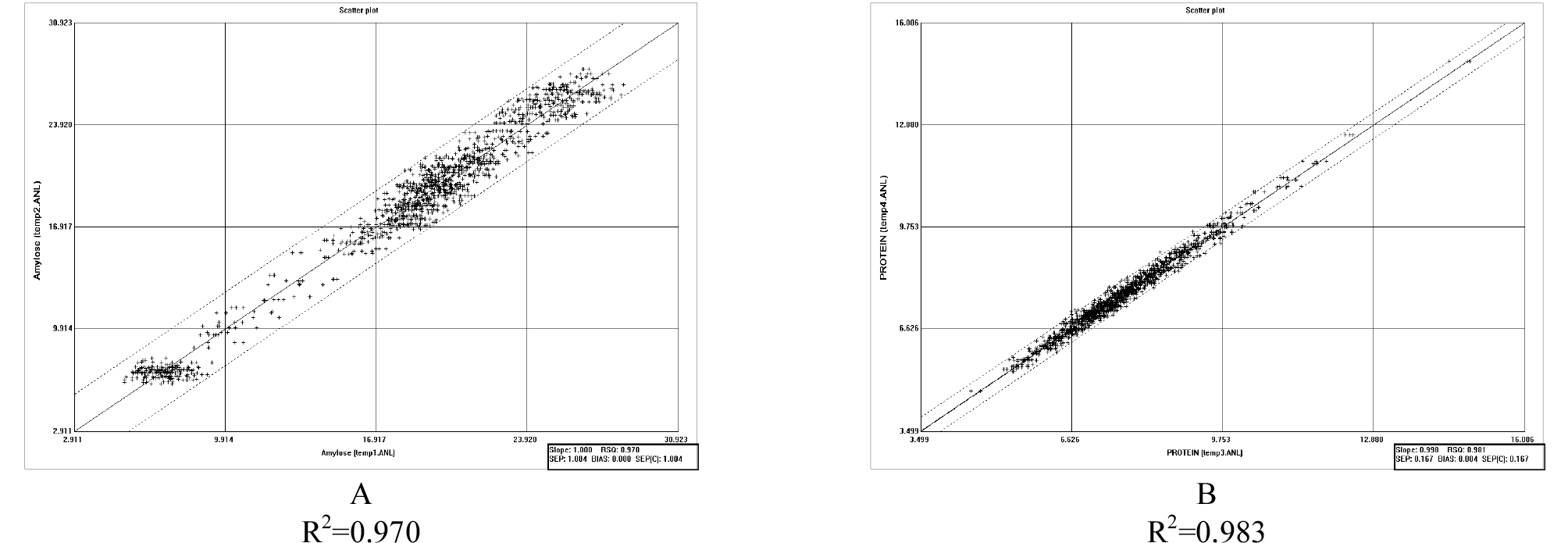
검량식 관련 주요 통계지표에는 RSQ (R2), 1-VR, SEC, SECV가 있다. RSQ는 NIRS 예측모델의 결정계수이고 1-VR은 교차검증 결정계수이다. 결정계수값이 1에 가까울수록 예측모델의 신뢰성은 높아진다. SEC는 검량식 표준오차이고 SECV는 교차검증 표준오차이다. 표준오차값이 0에 가까울수록 예측모델의 정확도는 높아진다. 작성된 검량식의 RSQ는 아밀로스에 있어 0.956이었고, 단백질은 0.982이었다. SEC는 아밀로스에 있어 1.159였고, 단백질은 0.164였다. 작성된 검량식의 정확도를 검증하기 위해 검량식 작성에 사용된 보존 자원을 사용하여 교차검증한 결과, 1-VR은 아밀로스에 있어 0.957이었고, 단백질은 0.981이었다. NIRS 분석방법은 습식분석에 비해 정확성은 낮기 때문에 검량식 자원들을 적정 농도 구간에서 균등한 자원 밀도를 나타내도록 구성하는 것이 중요하다(Kim et al., 2008). 자원밀도가 낮은 구간은 검량식 자원을 추가분석하여 보완하는 방법으로 NIRS 예측모델을 최적화하였다. 최적화된 NIRS 검량식과 관련 통계수치들을 나타냈다(Fig. 6, Table 8). 1-VR은 검량식 작성에 이미 이용된 자원을 재차 이용하여 정확도를 평가하는 역검정 방법이다. 간편하게 평가가 이뤄지는 장점이 있으나 1-VR 만으로는 개발된 NIRS 예측모델의 미지시료 분석 시 정확도를 평가하기에 부족하다(Bagchi et al., 2016). 따라서 검량식 작성에 사용된 자원 외의 별도자원들을 이용하여 NIR 기기내의 external validation 프로그램을 이용한 외부검증과정을 거쳤다. 검량식 작성에 사용된 511자원 이외의 재래종 2,000자원을 최적 예측모델이 적용된 NIRS를 이용하여 아밀로스 및 단백질 함량을 분석하였다. NIRS로 분석된 자원들을 저, 중, 고 함량 세 구간으로 나누고 각 함량의 구간별 대표자원을 선택하는 방법으로 아밀로스는 132자원, 조단백질은 124자원을 선정하여 NIRS 예측모델 검증자원으로 사용하였다. 선정된 검증자원들의 습식분석값을 NIRS 구동 프로그램의 lab data 항목에 입력하여 external validation set을 구성하였고, 최적 NIRS 예측모델에 external validation set이 적용된 검정 결과를 확인하였다(Table 8). 아밀로스와 단백질의 RSQ (r2) 값은 각각 0.962, 0.986이었고 SEP 값은 각각 2.349, 0.415였다. 단백질 SEP 값이 아밀로스 SEP 값보다 작으므로 단백질 측정 정확도가 높았으며 이는 아밀로스 SEP 0.882, 단백질 SEP 0.280 값으로 보고된 Hwang et al. (1994)의 결과와도 유사한 경향으로 나타났다. SEP 값이 아밀로스가 단백질에 비해 높게 나타나는 것은 현미에는 아밀로스와 관련이 적은 성분이 많이 존재하는 등 미강의 혼입 및 그 분산 상태가 백미에 비해 다르기 때문인 것으로 생각된다(Moon et al., 1994). 이상의 결과들을 종합해 볼 때 개발된 최적 NIRS 예측모델은 기존의 습식분석에 의한 성분 분석 결과와 높은 상관도를 가지며, 분석정확도 또한 큰 차이가 없는 것으로 나타났다(Oh et al., 2017a).
Table 8. External validation results of NIRS equation model for the amylose and protein content in the milled brown rice.
| Constituent | External validation | NIRS equation model | |||||||
| No. | r2 | SEP | Slope | No. | R2 | SEC | Slope | ||
| Amylose | 132 | 0.962 | 2.349 | 0.811 | 465 | 0.970 | 1.010 | 1.000 | |
| Protein | 124 | 0.986 | 0.415 | 1.038 | 473 | 0.983 | 0.158 | 0.998 | |
벼 유전자원의 집단분석
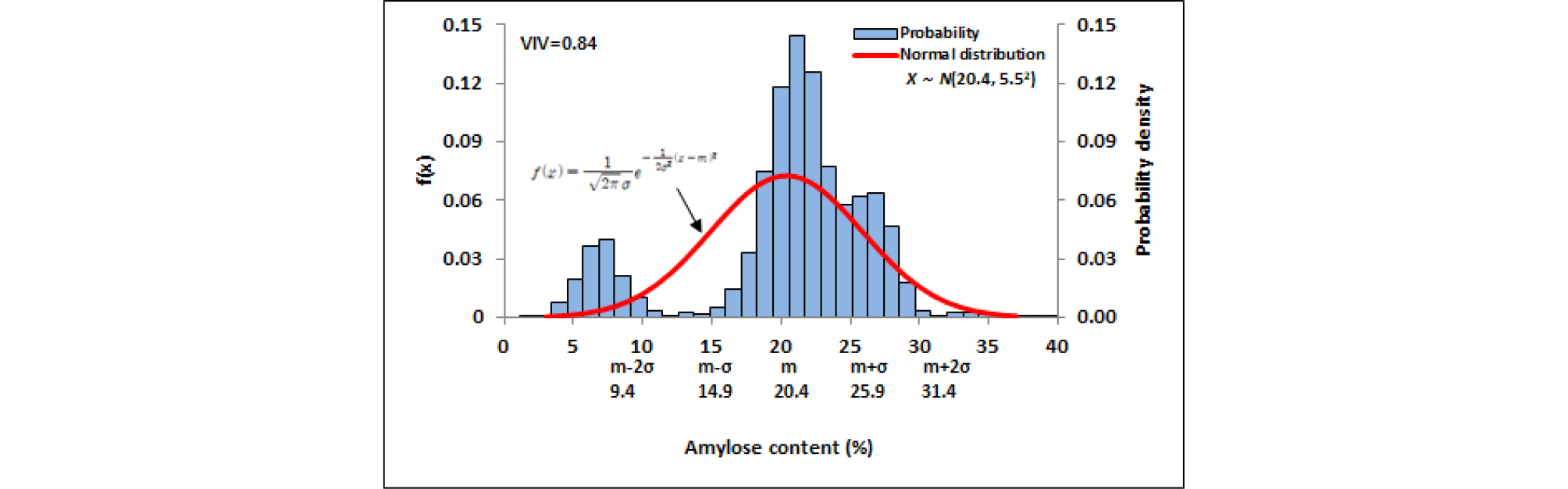
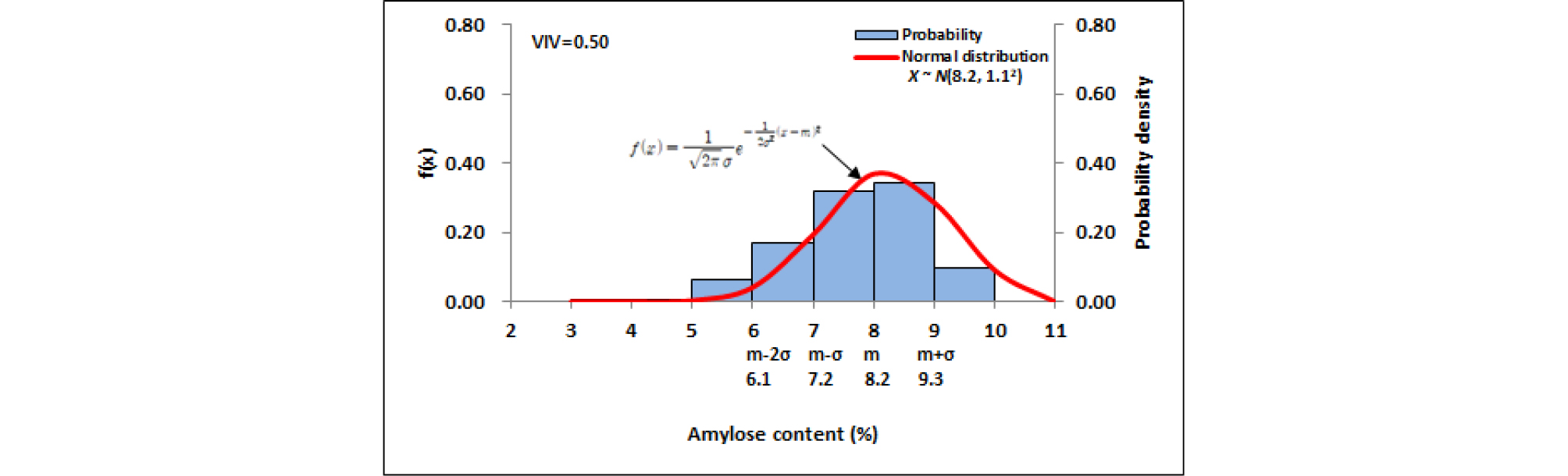
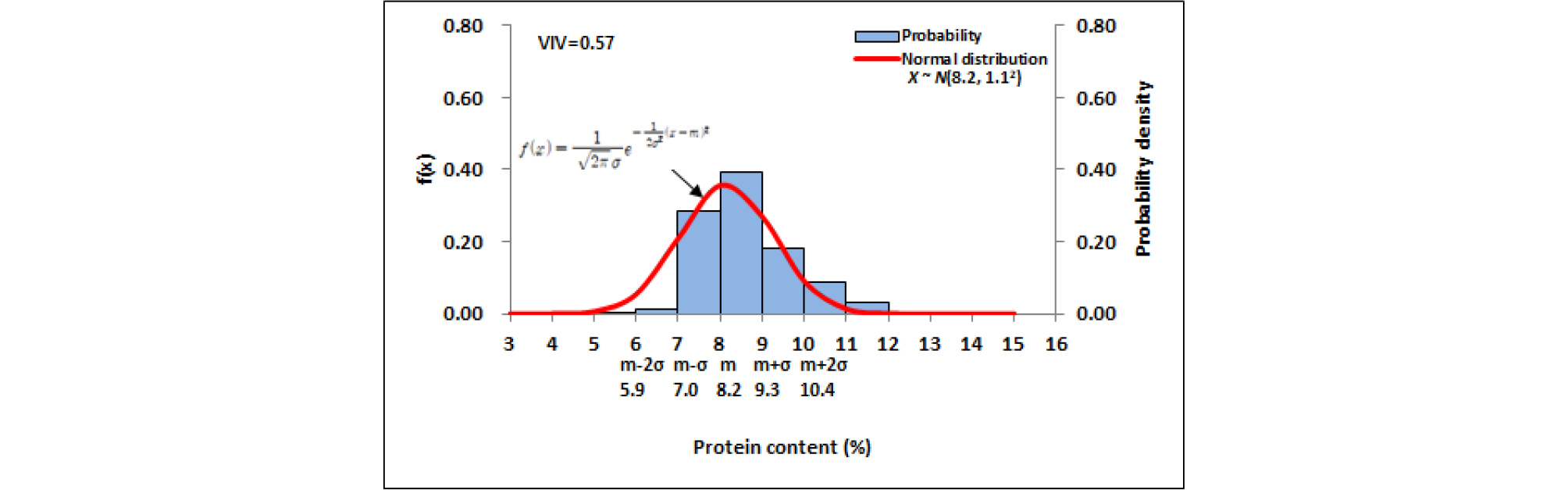
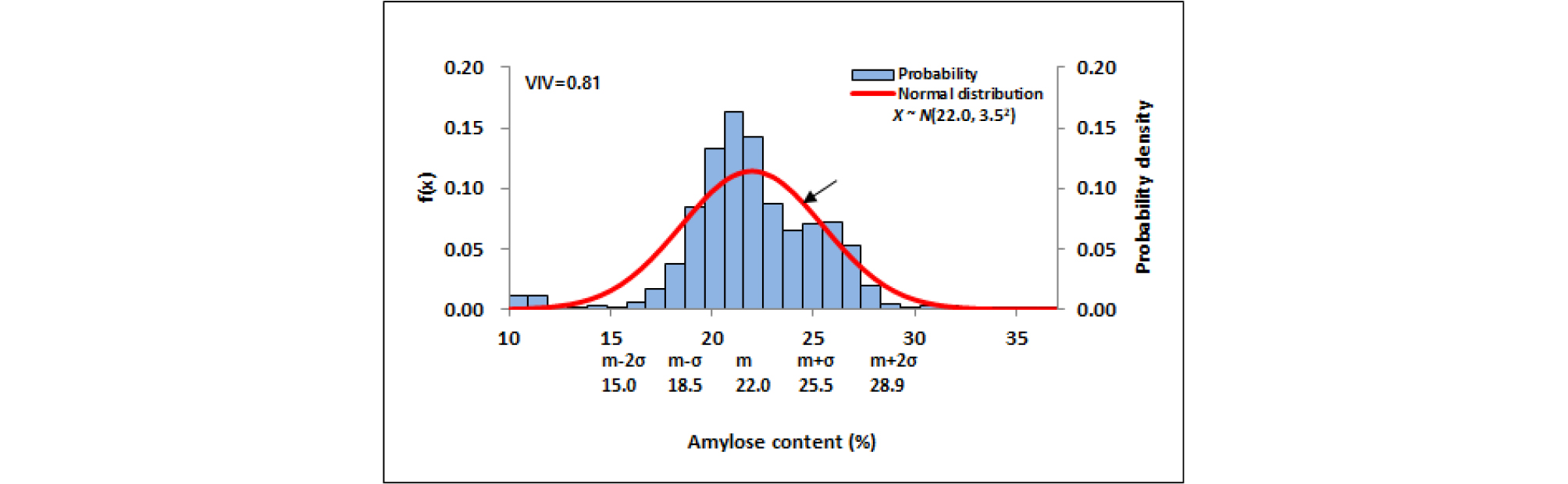
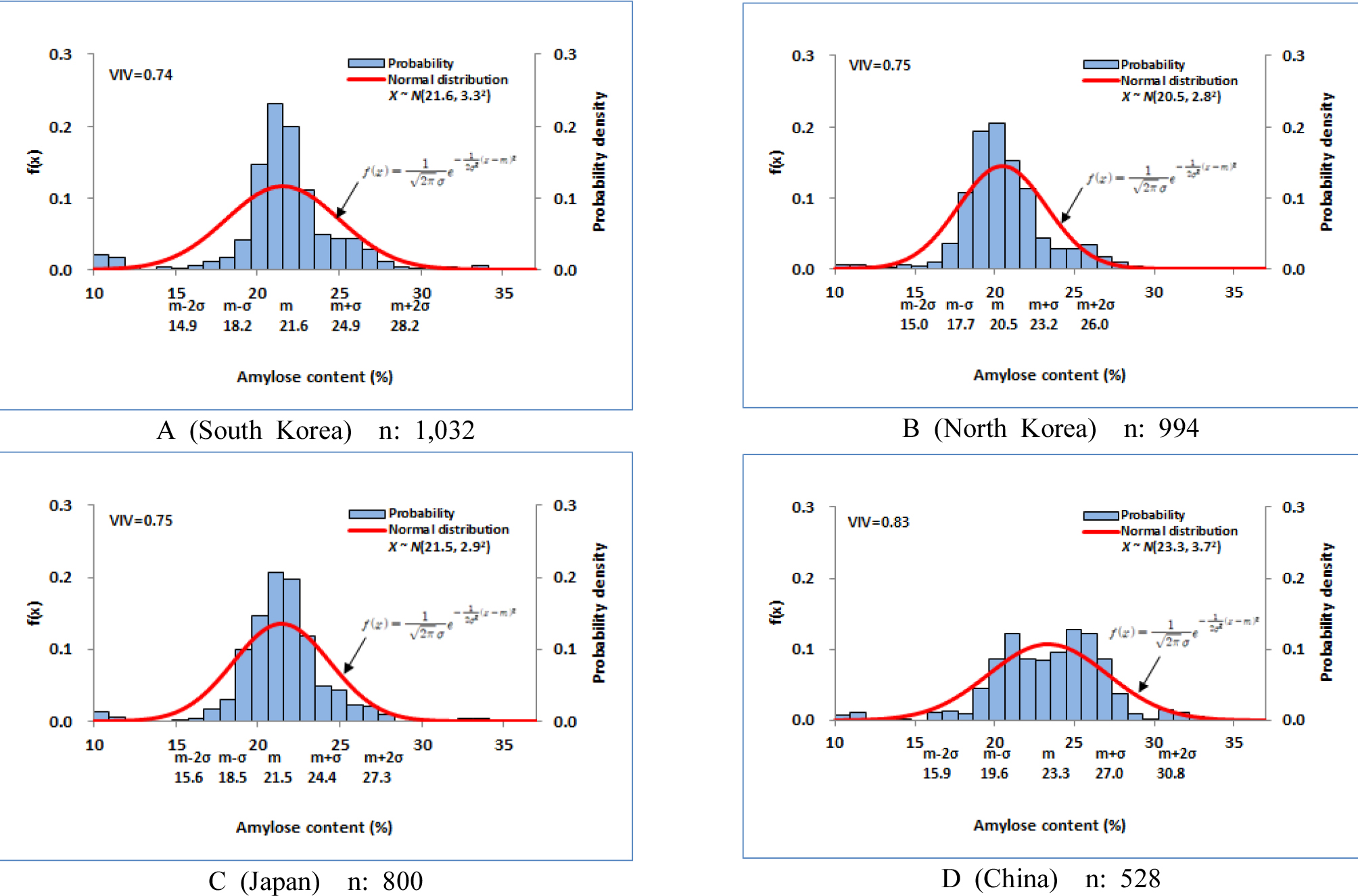
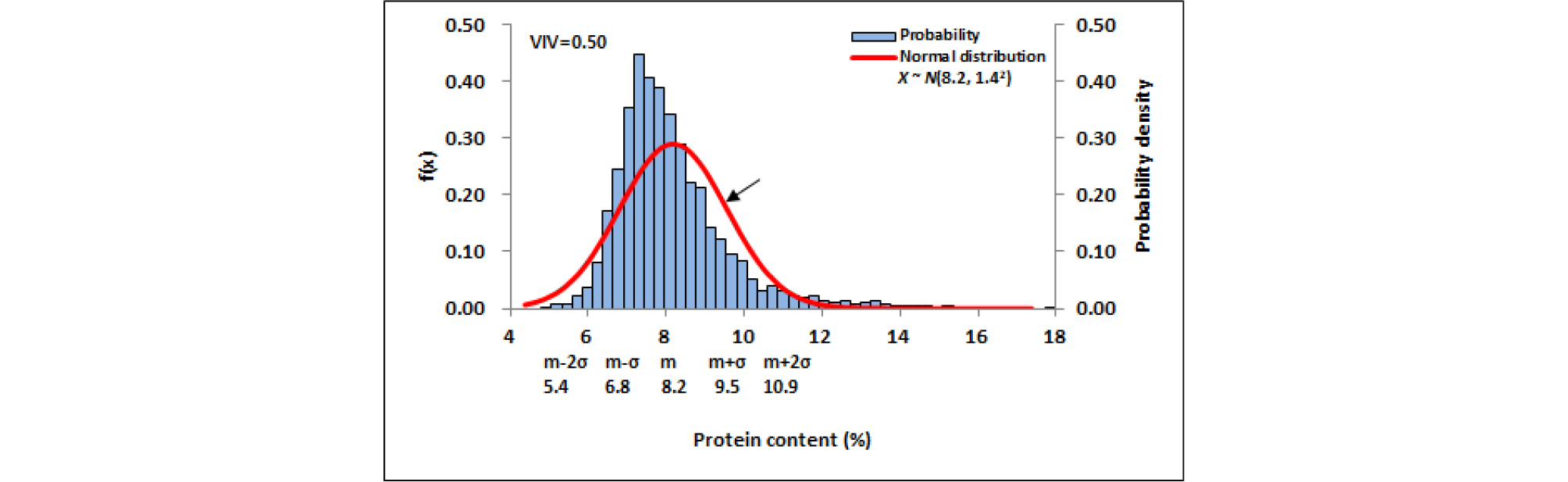
재래종 벼 4,948자원에 대한 아밀로스 함량분포의 정규분포함수에서는 확률밀도함수와 일치하지 않는 부분이 나타났으며, 정규분포곡선 속에서 뚜렷한 두 개의 집단으로 구분됨을 확인할 수 있었다. 이는 개략적으로 4.0-11.0% 구간과 14.0-35.0% 구간으로 대별되었다(Fig. 7). 두 개의 집단으로 구분되게 하는 아밀로스 함량구간은 11.0-14.0% 구간이었으며, 이 구간에는 수집자원이 거의 없었다. 이로 인하여 전체 아밀로스함량 분포가 정규분포를 이루지 않았다. Oh et al. (2017b)은 육종분야에서 벼 분류 기준으로 사용되는 아밀로스 함량범위(Jeong et al., 2013; Song et al., 2008; Kim et al., 1991)를 바탕으로 NIRS 측정 아밀로스 함량에 대하여 11% 이하를 찰벼, 11%이상을 메벼로 분류하였다. 이 분류기준과 국립식량과학원의 분류기준을 참고하여 본 연구에서는 아밀로스 함량 10.0% 이상 자원을 메벼로 분류하였고, 전체 정규분포에서 찰벼집단과 메벼집단으로 두 개의 정규분포 집단을 형성하였으나 자원분포가 많은 메벼 집단만을 대상으로 하여 함량 분포분석과 통계처리를 하였다. 재래종 벼 유전자원 4,948자원 중 찰벼 568자원의 아밀로스 함량별 자원분포는 Fig. 8과 같았으며, 9-10% 함량구간에서 자원밀도가 정규분포함수에 비해 상대적으로 낮았고, 평균 8.2%, 표준편차 1.1%, 다양성 지수는 0.50이었다. 찰벼 568자원의 단백질 함량별 자원분포는 Fig. 9와 같았으며, 5-7% 함량구간에서 자원밀도가 정규분포함수에 비해 상대적으로 낮았고, 평균 8.2%, 표준편차 1.1%, 다양성 지수는 0.57이었다. 메벼 4,380자원의 아밀로스 및 단백질 함량별 자원분포와 정규분포를 비교한 결과는 Fig. 10, Fig. 12와 같다. 메벼 자원은 아밀로스 15-18%과 23-24% 함량구간에서 자원밀도가 정규분포함수에 비해 다소 낮았으나, 전반적으로 자원분포는 평균값 22.0, 표준편차 3.5인 정규분포와 유사하였고, 자원의 다양성 지수는 0.81이었다(Fig. 10). 이와 같이 일부 구간에서 자원밀도가 매우 낮게 나타나는 것은 유전자원 수집이 부족하였거나 이에 해당하는 재래종 벼 자원이 없다는 의미로 판단된다. 41개국에서 수집된 재래종 메벼 4,380자원 중 한국 원산지 자원은 1,032자원, 북한 원산지 자원은 994자원, 일본 원산지 자원은 800자원, 중국 원산지 자원은 528자원으로 이들 자원들이 전체 재래종 자원의 76.6%를 차지하였다(Table 1). 국가별 자원분포분석에서 자원 비중이 큰 한국, 북한, 일본, 중국의 4개국 자원들을 대상으로 아밀로스 함량 분포특성을 상호 비교하였다(Fig. 11). 한국 원산지 자원은 아밀로스 15-19%와 24-25% 함량구간에서 정규분포함수에 비해 자원밀도가 낮았고, 20-22% 함량구간에서 정규분포함수에 비해 자원밀도는 높았다. 전반적으로 자원분포는 평균값 21.6, 표준편차 3.3인 정규분포와 유사하였고, 자원의 다양성 지수는 0.74였다(Fig. 11A). 북한 원산지 자원은 아밀로스 15-17%와 23-24% 함량구간에서 정규분포함수에 비해 자원밀도가 낮았고, 19-20% 함량구간에서 정규분포함수에 비해 자원밀도는 높았다. 전반적으로 자원분포는 평균값 20.5, 표준편차 2.8인 정규분포와 유사하였고, 자원의 다양성 지수는 0.75였다(Fig. 11B). 일본 원산지 자원은 아밀로스 15-18%와 24-26% 함량구간에서 정규분포함수에 비해 자원밀도가 낮았고, 21-22% 함량구간에서 정규분포함수에 비해 자원밀도는 높았다. 전반적으로 자원분포는 평균값 21.5, 표준편차 2.9인 정규분포와 유사하였고, 자원의 다양성 지수는 0.75였다(Fig. 11C). 중국 원산지 자원은 아밀로스 14-15%, 17-18%와 29-30% 함량구간에서 정규분포함수에 비해 자원밀도가 낮았고, 21-21.9%와 25-26% 함량구간에서 정규분포함수에 비해 자원밀도는 높았다. 전반적으로 자원분포는 평균값 23.3, 표준편차 3.7인 정규분포와 유사하였고, 자원의 다양성 지수는 0.83이었다(Fig. 11D). 한국, 북한, 일본 원산지 자원들의 아밀로스 함량분포는 상호 유사하였고, 중국 원산지 자원의 함량분포와는 달랐다. 아밀로스 다양성 지수는 한국, 북한, 일본 원산지 자원은 유사하였고, 중국 원산지 자원이 가장 높았다.
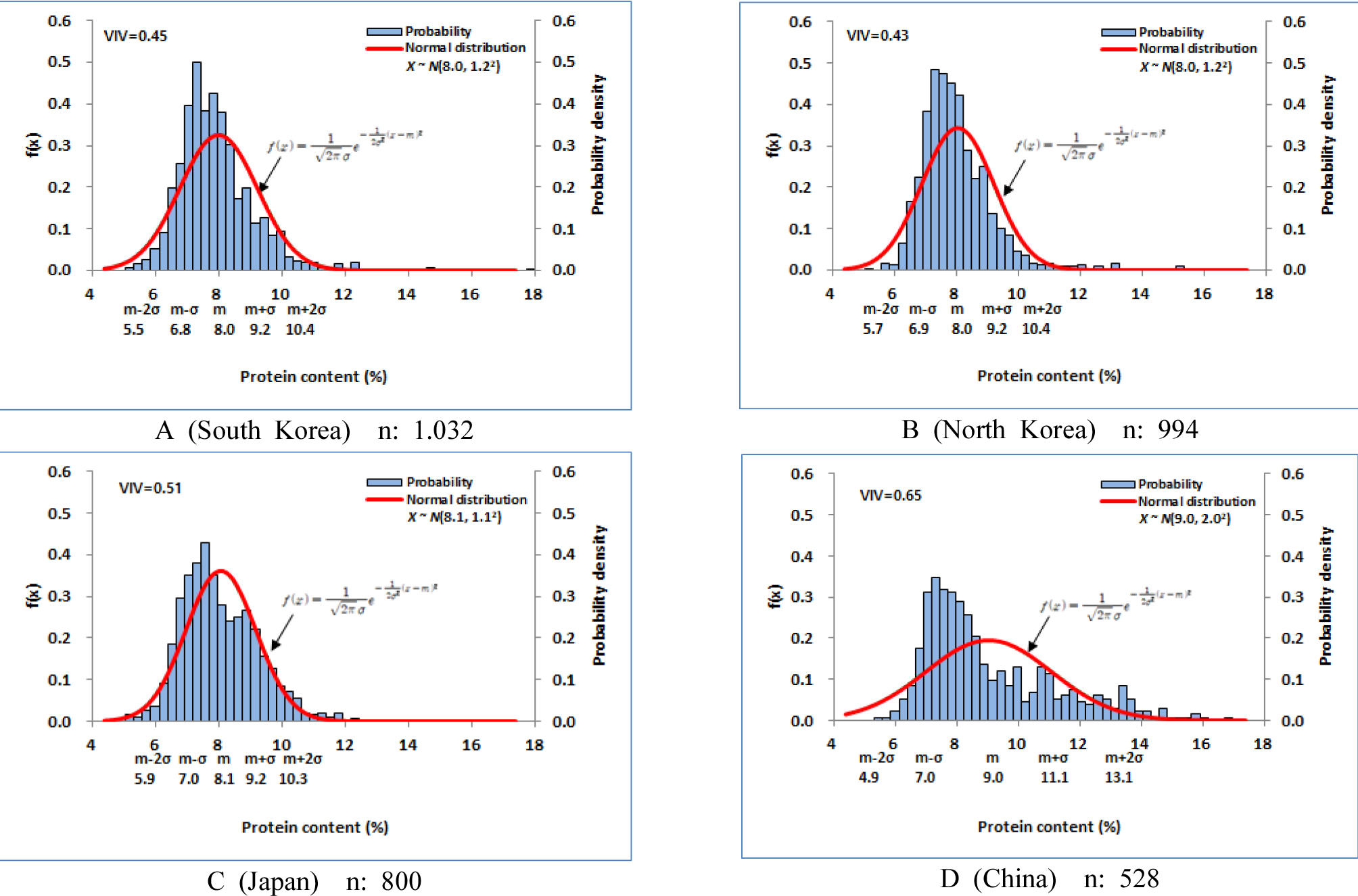
재래종 메벼 4,380자원의 단백질 함량분포를 나타냈다. 단백질 5-6%와 9-10.5% 함량구간에서 정규분포함수에 비해 자원밀도가 낮았고, 7-8% 함량구간에서는 정규분포함수에 비해 자원밀도는 월등히 높았다. 전반적으로 자원분포는 평균값 8.2, 표준편차 1.4인 정규분포와 유사하였고, 자원의 다양성 지수는 0.50이었다(Fig. 12). 국가별 자원분포분석에서 자원 비중이 큰 한국, 북한, 일본, 중국의 4개국 원산지 자원들을 대상으로 단백질 함량 분포특성을 상호 비교하였다(Fig. 13). 한국 원산지 자원은 단백질 8.5-9.0% 함량구간에서 정규분포함수에 비해 자원밀도가 낮았고, 7-8% 함량구간에서 정규분포함수에 비해 자원밀도는 높았다. 전반적으로 자원분포는 평균값 8.0, 표준편차 1.2인 정규분포와 유사하였고, 자원의 다양성 지수는 0.45였다(Fig. 13A). 북한 원산지 자원은 5-6.5% 함량구간에서 정규분포함수에 비해 자원밀도가 낮았고, 7-8% 함량구간에서 정규분포함수에 비해 자원밀도는 높았다. 전반적으로 자원분포는 평균값 8.0, 표준편차 1.2인 정규분포와 유사하였고, 자원의 다양성 지수는 0.43이었다(Fig. 13B). 일본 원산지 자원은 8-8.5% 함량구간에서 정규분포함수에 비해 자원밀도가 매우 낮았고, 6.5-7.5% 함량구간에서 정규분포함수에 비해 자원밀도는 높았다. 전반적으로 자원분포는 평균값 8.1, 표준편차 1.1인 정규분포와 유사하였고, 자원의 다양성 지수는 0.51이었다(Fig. 13C). 중국 원산지 자원은 단백질 4-6.5% 함량구간에서 정규분포함수에 비해 자원밀도가 낮았고, 7-8.5% 함량구간에서 정규분포함수에 비해 자원밀도는 다소 높았다. 전반적으로 자원분포는 평균값 9.0, 표준편차 2.0인 정규분포와 유사하였고, 자원의 다양성 지수는 0.65였다(Fig. 13D). 한국, 북한, 일본 원산지 자원들의 단백질 함량분포는 상호 유사하였고 중국 원산지 자원의 함량분포와는 달랐다. 단백질 다양성 지수는 북한 원산지 자원이 가장 낮았으며, 중국 원산지 자원이 가장 높았다. 히스토그램의 일정 계급폭(구간)으로 확률밀도함수를 구하면 계단모양의 확률밀도함수를 얻을 수 있다. 이를 바탕으로 하여 정규분포 함수로 그래프를 그리면 부드러운 곡선 형태를 이루게 되어 자원의 분포 값을 얻을 수 있다. 자원 집단의 확률밀도함수가 정규분포와 유사할 경우 정규분포의 표준화 과정을 통해 특정 함량구간의 확률 값을 구할 수 있고, 이 확률 값의 비율을 전체 자원수와 곱하면 특정 구간의 자원수를 계산할 수 있다. 이 표준정규분포 함수를 활용하여 정규분포에 적용하게 되면 자원관리를 정보화할 수 있는 등 자원 관리의 효율성을 높일 수 있을 것으로 사료된다.
벼 유전자원의 국가별 품종 기준(ANOVA)
독립변수 집단을 몇 개의 수준(또는 범주)으로 나누고 각 수준에 따라 나누어진 집단 간의 평균차를 검정하는 방법(차의 검정)으로써 서로 다른 세 개 이상의 집단에서 평균간 차이가 있는 지를 확인하고 자 할 때 분산분석이 이용된다. 독립변수는 아밀로스 또는 단백질 함량으로, 종속변수는 품종 재배의 기준점으로 정하여 국가별 품종 재배의 기준점이 상이한가를 검정해보고자 수집보존자원을 대상으로 통계분석을 실시하였다. 검정과정에서 필요한 유의성은 계산된 검정통계량(Fs)을 기각값(critical value, Fc)과의 크기 비교과정을 통해 결정된다. 이러한 과정은 정의된 수학공식에 관찰값과 관찰값 개수를 엑셀프로그램에 입력하면 비교적 용이하게 얻을 수 있다. 하지만 검정통계량의 개념과 그 개념의 기반이 되는 총제곱합, 군내제곱합, 군간제곱합을 이해하기에는 다소 어려움이 있으므로 이에 분산, 평균의 제곱, 제곱의 평균들 간의 상호관계를 도식(Fig. 3)으로 나타내어 설명하였고, 분산분석 절차를 순서도(Fig. 2)와 표(Table 3)로 정리하여 나타내는 한편 SST, SSB, SSW의 정의식과 계산식 유도과정을 정리하여 상호간의 관계를 보다 명확하게 제시하고자 하였다(식 (1)-식 (9)). 또한 공식과 개념의 나열에만 그치지 않고 실험 data를 근거로 분석방법을 적용한 예를 제시함으로써 실질적인 도움이 되고자 하였다(Table 9, Table 11). 재래종 메벼 4,380자원 중 높은 자원비율을 나타낸 한국 원산지 1,032자원, 북한 원산지 994자원, 일본 원산지 800자원, 중국 원산지 528자원을 대상으로 아밀로스 성분에 대한 분산분석을 실시한 결과 SSB 2,828.68, SSW 33,278.74를 얻었고, 또한 단백질 성분에 대한 분산분석 결과 SSB 444.90, SSW 6,072.16을 얻었다(Table 9, Table 11). 귀무가설(H0)은 ‘국가별 자원들의 함량 간 차이는 없다’로, 대립가설(H1)은 ‘국가별 자원들의 함량 간 차이는 있다’로 설정하였다. 아밀로스함량의 경우 검정통계량 Fs는 94.92였으며, 유의 수준 0.01, SSB 자유도 3, SSW 자유도 3,350일 때 F-분포표의 기각값(F0.01(3,3350))은 3.79이므로 검정통계량과 기각값의 비교에서 검정통계량의 값이 월등히 높으므로 귀무가설이 기각되고 대립가설이 채택되었다. 따라서 ‘벼 유전자원의 아밀로스 함량은 국가별 품종 기준이 다르다는 것을 수치화 할 수 있으며, 1% 유의수준에서 차이가 인정된다‘는 결론을 내릴 수 있었다(Table 10). 단백질 함량의 경우 검정통계량 Fs는 81.82, 유의 수준 0.01, SSB 자유도 3, SSW 자유도 3,350일 때 F-분포표의 기각값(F0.01(3,3350))은 3.79이므로 검정통계량과 기각값의 비교에서 검정통계량의 값이 월등히 높으므로 이 또한 귀무가설이 기각되고 대립가설이 채택되었다. 따라서 ‘벼 유전자원의 단백질 함량은 국가별 품종 기준이 다르다고 할 수 있으며, 1% 유의수준에서 차이가 인정된다’는 결론을 내릴 수 있었다(Table 12).
Table 9. Statistics data table for ANOVA test on non-glutinous rice amylose content based on geographical origin.
Table 10. ANOVA table of non-glutinous rice amylose contents in landrace rice germplasm from various countries.
| SV | df | SS | MSS | Fs | Critical value (F0.01(3,3350)) |
| SSB | 3 | 2828.68 | 942.89 | 94.92** | 3.79 |
| SSW | 3350 | 33278.74 | 9.93 | ||
| SST | 3353 | 36107.42 |
Table 11. Statistics data table for ANOVA test on non-glutinous rice protein content based on geographical origin.
Table 12. ANOVA table of non-glutinous rice protein contents in landrace rice germplasm from various countries.
| SV | df | SS | MSS | Fs | Critical value (F0.01(3,3350)) |
| SSB | 3 | 444.90 | 148.30 | 81.82** | 3.79 |
| SSW | 3350 | 6072.16 | 1.81 | ||
| SST | 3353 | 6517.06 |
벼 유전자원의 국가별 특성(DMRT)
분산분석 결과 집단 간 평균 간 유의미한 차이가 확인되면 어느 집단에서 유의한 차가 있는 지를 검정하여야 하며 이때 사용되는 통계방법 중 하나가 Duncan’s 다중범위검정(DMRT)이다. 이 방법은 각 집단들의 평균()과 LSR값을 비교하여 집단 간 평균차를 검정하는 방법이다. 와 LSR값의 차(│−LSR│)를 기준값으로 사용하여 다른 집단의 평균과 비교하거나 집단 간 평균 차(││)와 LSR값을 직접 비교하는 두 가지 방식을 사용할 수 있다. 이들 방법 간의 차이와 분석절차를 단계별 표(Tables 4-7)로 정리하여 나타냈고, DMRT 절차 순서도(Fig. 5)를 나타내는 등 시각적 표현을 통해 명확하게 제시하고자 하였다. 분산분석결과 한국, 북한, 일본, 중국 자원의 아밀로스 및 단백질 평균들 간 유의성이 인정되어 국가별 자원들 간의 유의차가 있는지를 Duncan’s 다중범위검정을 사용하여 확인하였다. 아밀로스 함량의 경우 계산된 는 0.11이었고, 군내 자유도 3,350, 유의수준 0.01일 때 SSR 값은 통계교과서에 수록된 ‘다중검정을 위한 스튜던트화한 범위’표를 참조하여 얻었다. 그 결과 계산된 LSR값과 아밀로스 평균함량 차의 값을 기준값으로 정하여 다중범위검정을 실시하였다. 아밀로스 분석의 경우 한국과 일본자원, 북한자원, 중국자원의 세 집단으로 나눌 수 있었으며, 국가별 평균 함량은 각각 한국 21.55%, 일본 21.45%, 북한 20.48%, 중국 23.34%였다. 중국자원의 DMRT 기준값(Amylose-LSR0.01)은 22.91이었고 나머지 세 자원들의 평균 아밀로스 함량 보다 크기 때문에 유의성이 있으며 결과적으로 중국자원은 독립집단을 이룬다고 할 수 있었다. 이에 대한 결과를 구체적으로 해석해 보면 중국자원은 한국, 일본의 자포니카 품종과는 구분되는 아밀로스 함량 특성을 나타낸 것으로 사료된다. 한국자원의 기준값이 21.13이고 일본자원 평균값이 21.45, 북한자원 평균값이 20.48이었므로(North Korea 20.48 < South Korea 21.13 < Japan 21.45) 한국자원과 일본자원은 유의성이 없고 한국자원과 북한자원은 유의성이 있으므로 한국자원과 일본자원은 동일 집단으로 간주되었고 북한자원은 한국자원과는 다른 독립집단을 이뤘다(Table 13). 단백질 함량의 경우 계산된 는 0.05였고, 군내 자유도 3,350, 유의수준 0.01일 때 SSR값은 ‘다중검정을 위한 스튜던트화한 범위표’를 참조하여 얻었다. 그 결과 계산된 LSR값과 단백질 평균함량 차의 값을 기준값으로 정하여 다중범위검정을 실시하였다. 단백질 분석의 경우 한국, 일본, 북한자원과 중국자원의 두 집단으로 나눌 수 있었으며, 국가별 평균 함량은 각각 중국 9.02%, 일본 8.06%, 북한 8.04%, 한국 7.99%였다. 중국자원의 DMRT 기준값(Protein-LSR0.01)은 8.82였고 나머지 세 자원들의 평균 단백질 함량 보다 크기 때문에 유의성이 있었으며 결과적으로 중국자원은 독립집단을 이룬다고 할 수 있었다. 일본자원의 기준값은 7.87이었고 북한자원의 평균값은 8.04, 한국자원 평균값은 7.99이었므로(Japan 7.87 < South Korea 7.99 < North Korea 8.04) 일본, 한국, 북한자원은 유의성이 없었고 동일 집단으로 간주되었다(Table 14). 중국자원의 아밀로스 평균함량은 23.34± 3.75%였고, 단백질은 9.02±2.05%였으며 다른 원산지 자원에 비해 가장 높은 수치를 나타냈다. 아밀로스와 단백질 함량에 있어 중국자원은 다른 원산지 자원들과는 구별되는 독립적인 집단을 구성하였다(Table 15). 한국자원의 아밀로스 평균함량은 21.55±3.34%였고, 단백질 평균함량은 7.99± 1.23%였다. 일본자원의 아밀로스 평균함량은 21.45±3.34%였고, 단백질 평균함량은 8.06±1.10%였다. 아밀로스와 단백질 함량에 있어 한국과 일본자원은 동일한 집단을 이뤘다(Table 15). 한국과 일본의 벼 품종은 대부분 자포니카이므로 이와 같은 결과는 상호 유사한 품종 특성이 반영된 것으로 사료된다. 일본의 경우 벼 품종 기준은 밥맛, 품질, 복합 내병성, 직파 적응성, 수량성의 5가지 항목에 중점을 두고 있으며 특히 밥맛검정 과정에서 NIRS를 이용하여 아밀로스, 단백질, 지방, 수분함량을 평가하고 있다(Park, 2005). 일본 소비자들은 아밀로스 함량이 낮은 벼 품종을 선호하기 때문에 일본자원의 품종 기준은 비교적 낮은 아밀로스와 낮은 단백질 함량에 있다고 사료된다.
Table 13. DMRT table of non-glutinous rice amylose content in landrace germplasm based on countries of origin.
Table 14. DMRT table of non-glutinous protein content in landrace rice germplasm based on countries of origin.
Table 15. Amylose and protein contents of non-glutinous in landrace rice germplasm based on countries of origin.
yMean separation within columns by Duncan's multiple range test at p=0.01.
적 요
본 연구는 선행연구에서 개발된 근적외선 분광분석(NIRS) 예측모델을 활용하여 측정된 국내외 재래종 메벼 유전자원의 아밀로스 및 단백질 함량 자료를 통계처리 하여 자원의 지리적 특성과 성분 함량에 대한 정확한 정보를 제공하기 위해 실시하였다.
1. 정규분포분석 결과 메벼 유전자원의 아밀로스 평균은 22.0%였고, 단백질 평균은 8.2%였으며 전체 자원의 95%를 차지하는 자원들의 함량범위는 아밀로스의 경우 15.0- 28.9%, 단백질은 5.4-10.9%였다. 자원의 다양성지수는 아밀로스의 경우 0.81, 단백질은 0.50이었다.
2. ANOVA, DMRT에 사용된 자원 수는 한국 자원의 경우 1,032, 북한은 994, 일본은 800, 중국은 528자원이었다. 국가별 아밀로스 평균함량은 중국 자원의 경우 23.34%, 한국 자원은 21.55%, 일본 자원은 21.45%, 북한 자원은 20.48%였다. 단백질 평균함량은 중국 자원의 경우 9.02%, 일본 자원은 8.06%, 북한 자원은 8.04%, 한국 자원은 7.99%였다. ANOVA 결과 벼 유전자원의 아밀로스 및 단백질 함량은 국가별 차이가 있었고 1% 유의수준에서 차이가 인정되었다.
3. DMRT 결과 국가별 아밀로스 함량은 한국과 일본, 북한, 중국의 세 집단으로 나눌 수 있었으며 각 집단 간 아밀로스 함량차이는 1% 유의수준에서 차이가 인정되었다. 단백질 함량의 경우 한국, 일본, 북한과 중국의 두 집단으로 나눌 수 있었으며 각 집단 간 단백질 함량차이는 1% 유의수준에서 차이가 인정되었다. 북한 자원은 가장 낮은 아밀로스 평균함량을 나타냈고, 한국 자원은 가장 낮은 단백질 평균함량을 나타냈다. 이에 비해 중국 자원은 가장 높은 아밀로스와 단백질 평균함량을 나타냈다. 이러한 지리적 분포에 따른 벼 자원 간 함량차이는 각 지역별 자원 선호도와 품종 특성이 반영된 결과라고 할 수 있다.