서 론
재료 및 방법
고시히카리×백일미 RIL집단 작물학적 특성평가
도열병 저항성 평가
고시히카리×백일미 RIL집단을 이용한 유전자지도 작성 및 QTL 분석
결 과
고시히카리×백일미 RIL집단 작물학적 특성평가
도열병저항성 QTL 분석
QTL 조합에 따른 도열병 저항성 변화
고시히카리×백일미 RIL 유망계통 선정
고 찰
적 요
서 론
벼는 국내외에서 매우 중요한 식량작물이지만 1인 가구의 증가와 서구화된 식생활 등으로 인해 우리나라 쌀 소비는 1980년대 이후 급격하게 감소(1인당 연간 쌀 소비량: ’89, 133.4 kg → ’19, 59.2 kg)하였다(KOSIS, 2020). 하지만 최근 3년간 그 감소추세가 둔화하고 있는데, 이는 한국인의 식단에서 쌀은 주식으로서 식생활에 매우 중요하다는 점을 시사한다(Kim, 2017). 따라서 고품질 밥쌀용 품종 개발은 지속적으로 수행되어야 한다.
‘맛있는 쌀’로 브랜드 인지도가 높은 일본 도입종 ‘고시히카리’는 국내에서 매우 인기 있는 외래품종이고(Mo et al., 2019), 육성된 지 60년이 넘었지만 일본에서도 가장 많이 재배되는 품종이다(Kobayashi et al., 2018). 하지만 고시히카리는 도열병에 취약하고 키가 커서 잘 쓰러지는 치명적인 단점이 있고, 이를 육종적으로 보완하기 위하여 일본 주산지인 니카타현을 중심으로 고시히카리 형질을 최대한 유지하면서 도열병, 쓰러짐에 강한 근동질 계통을 꾸준히 육성하고 있다(Ishizaki et al., 2005). 특히 고시히카리의 유전적 배경에 다양한 도열병 저항성 유전자를 도입한 근동질 계통인 고시히카리 BL (Blast Resistance Lines) 품종의 경우 시장에서 원품종인 고시히카리와 구분 없이 유통되고 있다(Kobayashi et al., 2018).
도열병(Magnaporthe grisea)은 벼를 재배하는 모든 지역에서 생육초기에서 후기까지 모든 생육시기에 큰 피해를 주는 대표적인 병이다(Li et al., 2007). 벼 도열병 방제에 여러 방법이 있으나, 가장 경제적이고 효과적인 방법은 저항성 품종을 활용하는 것으로 알려져 있다(Khush & Jena, 2009). 농촌진흥청 국립식량과학원에서 육성한 ‘백일미’는 도열병 저항성이 우수하고 이앙 후 100일 이내에 수확이 가능한 극조생종이다(Mo et al., 2019). 본 연구는 우수한 밥맛을 지닌 고시히카리에 도열병에 강한 백일미를 교배하여 육성한 RIL (recombinant inbred line) 집단에서 도열병 저항성에 대한 유전분석을 수행함으로써 1) 도열병 저항성 유전자의 염색체 상 위치를 파악하고 각 유전자위의 작용가를 추정하며, 2) 고시히카리의 우수한 미질을 보유하면서 도열병에도 강한 계통을 선발하기 위하여 수행하였다.
재료 및 방법
고시히카리×백일미 RIL집단 작물학적 특성평가
고시히카리×백일미 RIL집단(394계통) 및 모·부본은 2019년 국립식량과학원 작물육종과(전북 전주)에 공시하여 작물학적 특성을 조사하였다. 보통기 재배에 적합하게 파종은 2019년 5월 5일 실시하여 31일간 육묘한 후 6월 5일에 이앙하였고, 농촌진흥청 표준 재배법을 준수하여 재배하고 농업과학기술 연구조사분석기준(RDA, 2011)에 의거하여 출수기, 간장, 수장, 수수를 조사하였다. RIL 394계통의 출수기, 간장, 도열병 저항성 검정 성적을 기준으로 1) 출수기와 간장이 고시히카리와 유사하면서 도열병에 강한 계통(KR), 2) 출수기와 간장이 고시히카리와 유사하면서 도열병에 약한 계통(KS), 3) 출수기와 간장이 백일미와 유사하면서 도열병에 강한 계통(BR)을 각각 15계통씩 선발하여 추가적으로 현미 천립중, 장·폭 및 완전립률(%)을 조사하였다. 현미 장·폭 및 완전립률(%) 측정에는 Rice Quality Analyzer (RN300 model, Kett, Japan)을 이용하였다.
도열병 저항성 평가
도열병 저항성 검정은 2019년 국립식량과학원 작물육종과(전북 전주)와 운봉시험지(전북 남원)에 공시하여 잎 도열병 발병 정도를 검정하였다. 이병성 품종으로는 호평벼를 활용하여, 전주와 남원에 각 2019년 7월1일, 6월 24일에 파종하였고, 잎 도열병 이병정도(1~9)를 각각 8월 9일, 8월 7일에 농업과학기술 연구조사분석기준(RDA, 2011)에 의거하여 조사하였다(1: 강, 9 : 약).
고시히카리×백일미 RIL집단을 이용한 유전자지도 작성 및 QTL 분석
고시히카리×백일미 RIL집단의 유전자지도를 작성하기 위하여 최초에 F1 3개 식물체로부터 유래한 총 394계통의 RIL 중 단일 F1 식물체에서 유래한 142계통의 RIL을 임의로 선정하여 유전자형을 분석하였으며, 제한효소 ApeKI를 활용한 GBS (Genotyping By Sequencing)를 통하여 분석하였다(Jang et al., 2019). GBS를 통하여 탐지한 875개 SNP (single nucleotide polymorphism) 중 142계통에서 유전자형 결측치가 20%를 초과하는 129개 SNP와 분리비 이상(segregation distortion)을 보이는 237개 SNP를 제외한 509개를 유전자지도 작성에 이용하였다. 이 중 유전적 위치가 중복되는 SNP를 제외하고 최종적으로 130개 SNP로 이루어진 유전자지도를 작성하였다.
양적유전자좌(QTL)분석에는 QTL IciMapping V4.1 프로그램을 활용하였다(Meng et al., 2015). QTL 분석은 도열병 저항성 검정 표현형 데이터와 SNP 유전자형 데이터를 이용하여 IM (Interval Mapping) 방법과 CIM (Composite Interval Mapping) 방법으로 수행하였다(Zhang et al., 2008). 유의한 QTL을 결정하기 위한 LOD 임계값은 p<0.05 조건에서 1,000회 순열검정(permutation test)하여 결정하였다(Churchill & Doerge, 1994). QTL 명명은 McCouch (2008) 방법을 준수하였다(McCouch, 2008).
IM과 CIM에서 모두 유의한 것으로 탐지된 염색체 1번과 2번 상의 QTL에 대하여 RIL 394계통의 유전자형을 결정하기 위하여 각 QTL의 peak에 근접한 부위에서 CAPS (Cleaved Amplified Polymorphic Sequence) 분자표지를 작성하였다. 분자표지 작성에는 국립식량과학원 작물육종과에서 보유중인 고시히카리와 백일미 염기서열 데이터를 활용하여 두 품종 간 서열 차이를 보이는 SNP를 선정하였으며, 분자표지 정보는 다음과 같다. 1번 염색체 상의 QTL에 활용된 CAPS마커의 프라이머 서열은 forward 5’-TCGGG GGCTTGCGATCTGCT-3, reverse 5’-TCAGCGGAGATC ATGTGGTTGAGC-3’이었고 제한효소 Hhal를 활용하여 백일미와 고시히카리 유전자형을 구분하였다. 2번 염색체 상의 QTL에 활용된 CAPS 마커의 프라이머 서열은 forward 5’-GCTGCCTGCCTGTCCCTAGC-3’, reverse 5’-TGGCT GCTAGTATCTGGTTGACGC-3’이었고 제한효소 BspDI를 활용하여 백일미와 고시히카리 유전자형을 구분하였다. PCR 조건은 처음 denaturation은 95°C에서 5분, 그리고 95°C에서 20초 denaturation, 58°C에서 30초 annealing, 72°C에서 90초 extension 한 후, 이를 40회 반복한 후, 마지막으로 72°C에서 10분 extension 으로 수행되었다. PCR 산물을 제한효소 처리한 후 1.5% 아가로스젤에 전기영동하여 밴드 패턴을 확인하였다.
결 과
고시히카리×백일미 RIL집단 작물학적 특성평가
KBRIL (Koshihikari × Baegilmi RIL population, n=394)을 원품종인 고시히카리, 백일미와 함께 공시하여 주요 작물학적 특성을 평가하였다(Table 1). 고시히카리의 출수일수는 94일로 백일미보다(82일) 약 12일가량 늦었고, 간장은 각 76 cm, 71 cm로 고시히카리가 백일미보다 5 cm 길었으며, 수장 및 수수는 두 품종 모두 22 cm, 11개로 동일하였다. 도열병 저항성 정도는 고시히카리는 매우 약(9)하고, 백일미는 매우 강한(1) 저항성을 보였다(Fig. 1).
Table 1.
Main agronomic traits of the Koshihikari × Baegilmi recombinant inbred line population (KBRIL).
KBRIL (n=394)의 출수일수, 간장, 수장, 수수 범위는 각 61~119일, 47~92 cm, 14~25 cm, 7~17개로, 4개 형질 모두 양친의 범위를 크게 벗어났다(Table 1). 도열병 저항성 분포는 지역별로 저항성(1~3), 중도저항성(4~6), 감수성(7~9)으로 나눌 때 전주에서는 저항성 189계통(48%), 중도저항성 127계통(32%), 감수성 78계통(20%)으로 분포하였다. 남원에서는 저항성 230계통(58%), 중도저항성 73계통(19%), 감수성 91계통(23%)의 분포를 보였다.
도열병저항성 QTL 분석
고시히카리×백일미 RIL집단의 유전분석은 GBS 분석이 이루어진 142계통을 활용하여 130개 SNP 마커로 연관지도를 작성하였다. 연관지도의 총 길이는 1272.7 cM이었으며, 마커 간의 평균거리는 9.9 cM이었다(Fig. 2). 염색체당 마커의 수는 6~14개이었으며, 염색체당 평균 11개 마커가 분포하였다. 전주와 남원 각 지역의 도열병 저항성 검정결과에 대하여 IM, CIM 분석으로 QTL을 탐색한 결과는 아래와 같다.
전주의 경우 IM, CIM 분석에서 유의한 QTL 선정을 위한 LOD 임계값은 각각 7.5, 7.2로, 1번 염색체 상의 S1_ 30019984 (106.42 cM)와 S1_34790922 (117.51 cM) 사이의 영역이 동일하게 탐지되어 qBL1.1로 명명하였다(Table 2, Fig. 3). 전주에서 qBL1.1의 LOD 값은 IM과 CIM에서 모두 13.8이고, 표현형 변이 설명률은 각 15.9%, 36.0%이며, 상가적 효과는 1.2로 동일하였다. 한편 IM 분석에서 11번 염색체 상에 있는 비교적 넓은 영역(28.10~40.75 cM)의 QTL이 탐지되었으나, CIM 분석에서는 탐지되지 않았기에 추후 분석에서는 제외하였다.
Table 2.
Quantitative trait loci (QTL) for blast resistance identified from the Koshihikari × Baegilmi recombinant inbred line (RIL) population (n = 142).
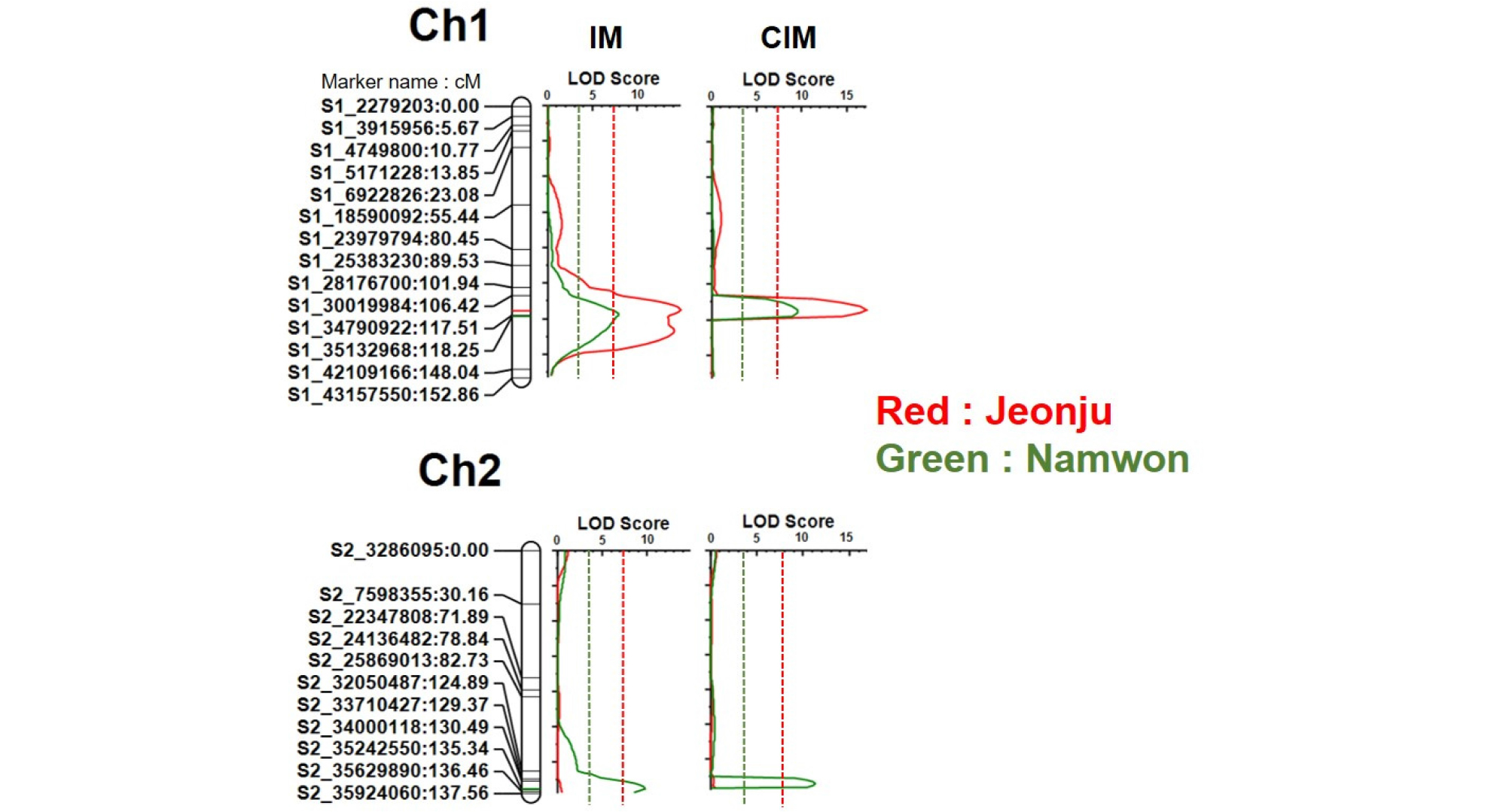
Fig. 3.
Blast resistance quantitative trait loci detected on chromosomes 1 and 2 in the Koshihikari × Baegilmi recombinant inbred line population (n = 142). Dotted lines indicate the logarithm of odds (LOD) thresholds in Jeonju and Namwon determined by 1,000 permutations. IM: interval mapping, CIM: composite interval mapping.
남원의 경우 IM, CIM 분석에서 유의한 QTL 선정을 위한 LOD 임계값은 각각 3.4, 3.2로, 1번 염색체 상에서 IM분석에서는 S1_34790922 (117.51 cM)와 S1_35132968 (118.25 cM) 사이가, CIM 분석에서는 S1_30019984 (106.42 cM)와 S1_34790922 (117.51 cM) 사이가 각각 유의한 QTL로 탐지되어 전주에서 탐색된 qBL1.1과 겹치는 위치였다(Table 2, Fig. 3). 남원에서 qBL1.1의 LOD값은 IM과 CIM에서 각각 7.8, 8.8이고, 표현형 변이 설명률은 각 20.7%, 20.5%였으며, 상가적 효과는 각 1.1, 1.0이었다. 또한 남원에서는 IM, CIM 분석에서 모두 2번 염색체 상의 S2_34000118 (130.49 cM)과 S2_35242550 (135.34 cM) 사이의 영역이 유의한 QTL로 탐지되어 이를 qBL2.1로 명명하였다. qBL2.1의 LOD 값은 IM과 CIM에서 각 9.7, 11.3이었고, 표현형 변이 설명률은 각 22.3%, 24.7%, 상가적 효과는 각 1.2, 1.1이었다.
결론적으로 전주에서는 1번 염색체에 위치한 도열병저항성 QTL qBL1.1이, 남원에서는 1, 2번 염색체에 위치한 도열병저항성 QTL qBL1.1, qBL2.1이 탐지되었다(Fig. 3, Table 2).
QTL 조합에 따른 도열병 저항성 변화
두 개의 도열병 저항성 QTL (qBL1.1, qBL2.1) 대립인자 조합에 따른 집적효과를 확인하였다. KBRIL 394계통을 qBL1.1과 qBL2.1의 고시히카리(K), 백일미(B) 대립인자 여부에 따라 총 4개의 그룹으로 나누었고, 이를 qBL1.1_K+ qBL2.1_K (n=95), qBL1.1_B+qBL2.1_B (n=101), qBL1.1_K+ qBL2.1_B (n=98), qBL1.1_B+qBL2.1_K (n=86)로 구분하였다(Fig. 4).

Fig. 4.
The distribution of blast resistance and other agronomic traits of the four groups carrying different alleles at qBL1.1 and qBL2.1. Different letters (a, b, c) indicate significant differences according to Tukey’s HSD tests (ns : not significant). Error bars indicate standard deviations. JJ: Jeonju, NW: Namwon, K: Koshihikari, B: Baegilmi.
전주에서 4개 그룹의 도열병 저항성 평균은 qBL1.1_K+ qBL2.1_K가 6.7, qBL1.1_B+qBL2.1_B가 3.0, qBL1.1_K+ qBL2.1_B가 5.1, qBL1.1_B+qBL2.1_K가 3.2이었다. 전주에서는 두 QTL에 고시히카리 대립인자를 보유한 그룹(qBL1.1_K+qBL2.1_K)에 비하여 두 QTL에 백일미 대립인자를 보유하거나(qBL1.1_B+qBL2.1_B) 하나에만 백일미 대립인자를 보유한 그룹(qBL1.1_K+qBL2.1_B, qBL1.1_B+ qBL2.1_K)이 모두 도열병 저항성이 유의하게 강하였다. 하나의 QTL에서만 백일미 대립인자를 보유한 경우, qBL1.1에 백일미 대립인자를 보유한 그룹이 qBL2.1에 백일미 대립인자를 보유한 그룹에 비해 도열병 저항성이 유의하게 강하였다. qBL1.1_B+qBL2.1_B는 qBL1.1_K+qBL2.1_B에 비하여 도열병 저항성이 유의하게 강하였으나 qBL1.1_B+ qBL2.1_K와는 유의한 차이가 없어 qBL2.1의 백일미 대립인자 집적에 의한 도열병 저항성 강화 효과는 없는 것으로 판단되었다.
남원에서 QTL 대립인자 조합에 따른 4개 그룹의 도열병 저항성 평균은 qBL1.1_K+qBL2.1_K가 8.1, qBL1.1_B+ qBL2.1_B가 2.6, qBL1.1_K+qBL2.1_B가 3.6, qBL1.1_B+ qBL2.1_K가 3.7로, 두 QTL 모두에 백일미 대립인자를 보유한 그룹이 가장 강한 저항성을 보였다. 하나의 QTL에서만 백일미 대립인자를 보유한 경우(qBL1.1_K+qBL2.1_B, qBL1.1_B+qBL2.1_K)에는 두 QTL 모두에 고시히카리 대립인자만 보유한 그룹에 비해 도열병 저항성이 유의하게 강하였으나, 두 QTL 모두 백일미 대립인자를 보유한 그룹보다는 유의하게 약하였다. 따라서 전주에서와는 달리 qBL1.1, qBL2.1 모두 백일미 대립인자가 집적될 때 도열병 저항성이 강화되는 것을 볼 수 있었다(Fig. 4).
대립인자 조합에 따른 4개 그룹(qBL1.1_K+qBL2.1_K, qBL1.1_B+qBL2.1_B, qBL1.1_K+qBL2.1_B, qBL1.1_B+ qBL2.1_K)의 평균 출수일수는 각각 88.9, 87.8, 87.7, 88.5일, 간장은 70.4, 69.8, 69.2, 69.5 cm, 수장은 20.9, 20.9, 20.7, 20.5 cm, 수수는 10.5, 10.3, 10.2, 10.6개로 4개 형질 모두 유의한 차이를 보이지 않았다. 따라서 qBL1.1, qBL2.1은 출수일수, 간장, 수장, 수수에 영향하지 않으면서 도열병 저항성을 향상시키기 위하여 활용할 수 있는 유용한 QTL로 판단된다.
고시히카리×백일미 RIL 유망계통 선정
KBRIL 394계통 중에서 출수기, 간장, 도열병 저항성을 기준으로 1) 출수기와 간장이 고시히카리와 유사하면서 도열병에 강한 계통(KR), 2) 출수기와 간장이 고시히카리와 유사하면서 도열병에 약한 계통(KS), 3) 출수기와 간장이 백일미와 유사하면서 도열병에 강한 계통(BR)을 각각 15계통씩 선발하여 그룹화하였다. 출수기 선정범위는 파종 후 출수기까지 누적평균일조시간(AH) 및 누적평균온도(AT)을 고려하였고, 간장범위는 양친의 ±5 cm이었다(Table 3, Fig. 5).
Table 3.
Criteria for selecting the three different recombinant inbred line (RIL) groups (KR, KS, and BR).
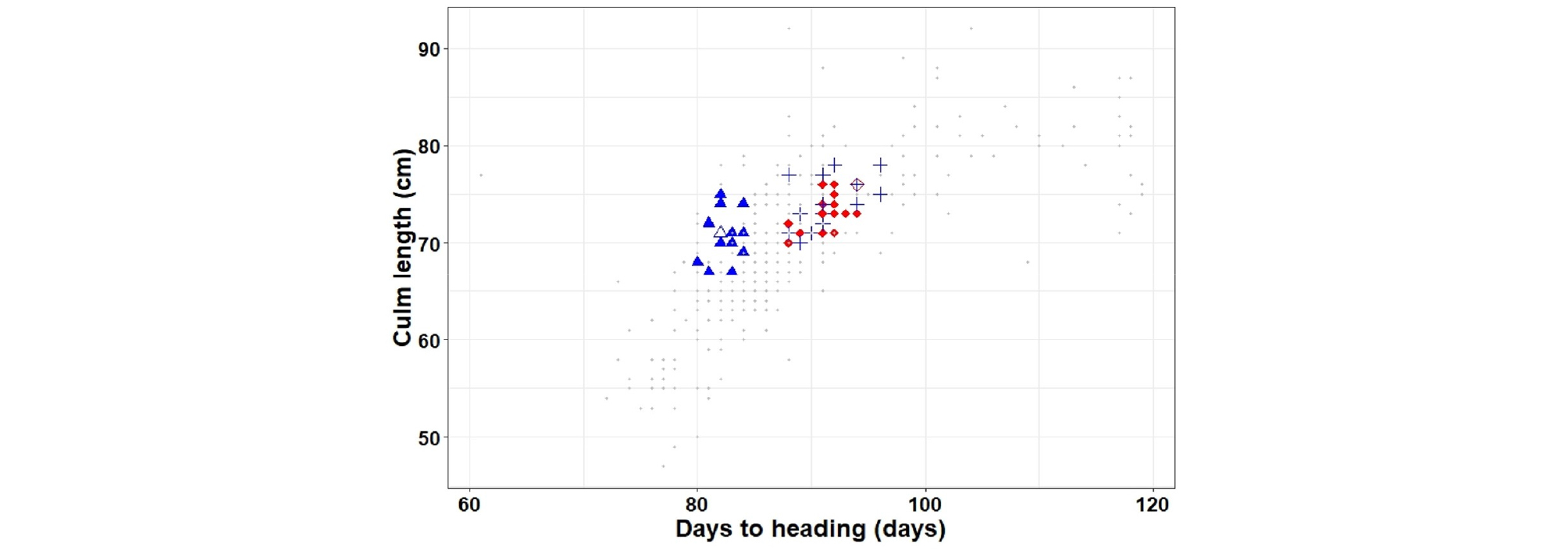
Fig. 5.
Days to heading and culm length of the three recombinant inbred line groups (KR, KS, and BR). The three groups are represented by different shapes (◇ : Koshihikari, △ : Baegilmi, + KR: Koshihikari-type RILs with blast resistance, ◆ KS: Koshihikari-type RILs with blast susceptibility, ▲ BR: Baegilmi-type RILs with blast resistance).
고시히카리의 출수기(전주, 8월 7일), AH (662시간), AT (2,172°C)를 고려하여 8월 1~9일에 출수하고, 간장이 70~80 cm인 고시히카리와 유사한 계통을 선발하였다. 이들 중 전주와 남원에서 모두 도열병에 강한(1~3) 15계통(KR), 도열병에 약한(4~9) 15계통(KS)을 선발하였다. 또한 백일미의 출수기(전주, 7월 26일), AH(576시간), AT(1,829°C)를 고려하여 출수기가 7월 23~28일이면서, 간장이 67~75 cm 범위이고, 전주와 남원에서 모두 도열병에 강한 15계통(BR)을 선발하였다(Table 3).
3개 그룹(KR, KS, BR)의 도열병 저항성, 출수일수, 간장, 수장, 수수, 현미 천립중, 현미 길이, 현미 폭, 현미 완전립률(%)을 비교하였다(Table 4). 출수일수는 KR그룹과 KS그룹은 91일로 동일하였고, BR그룹은 82일로 출수가 유의하게 빨랐다. 간장은 KR, KS, BR그룹 각 76 cm, 73 cm, 70 cm으로 KR과 KS에 비하여 BR이 유의하게 작았다. 수장은 3그룹 모두 22 cm로 동일하였다. 수수는 KR, KS는 10개, BR은 12개로 유의한 차이를 보이지 않았다. 천립중은 KR과 KS는 둘 다 19.5 g, BR은 20.1 g으로써 역시 유의한 차이를 보이지 않았다. 현미 길이와 폭은 세 그룹 모두 각각 5.1 mm, 2.8 mm로 유의한 차이가 없었다. 하지만 KR의 현미 완전립률(55%)은 KS (46%), BR (47%)에 비해 유의하게 높았다. 결론적으로 KR, KS, BR 3개 그룹은 도열병, 출수일수, 간장, 현미 완전립률을 제외하고는 조사된 형질에 유의한 차이를 보이지 않았다.
Table 4.
Important agronomic traits of the three recombinant inbred line (RIL) groups (KR, KS, and BR).
고 찰
본 연구는 고품질 밥쌀용 품종으로 인지도가 높지만 도열병에 취약한 고시히카리와 도열병에 강하면서 생육이 빠른 백일미를 교배하여 만든 RIL집단(KBRIL)에서 도열병 저항성에 대한 유전분석을 수행하여 저항성 유전자의 염색체 상 위치를 규명하고, 고시히카리의 우수한 미질을 보유하면서 도열병에도 강한 계통을 선발하기 위해 수행되었다.
KBRIL로부터 도열병 저항성 QTL qBL1.1과 qBL2.1를 각각 1번, 2번 염색체 상에서 탐색하였다. qBL1.1, qBL2.1은 출수일수, 간장, 수장, 수수에 영향을 주지 않아 주요 농업 형질에 영향을 주지 않으면서 도열병 저항성을 향상시키기 위하여 활용할 수 있는 유용한 QTL로 판단된다. 두 QTL의 백일미 대립인자 집적효과는 지역에 따라 상이하였다. 전주에서는 qBL1.1에서만 백일미 대립인자의 집적에 의한 도열병 저항성 강화 효과가 확인되었지만, 남원에서는 qBL1.1과 qBL2.1 모두 백일미 대립인자가 집적될 때 도열병 저항성이 향상됨을 확인하였다. 이는 2번 염색체 상의 qBL2.1이 두 지역 중 남원에서만 유의한 QTL로 탐지된 것과 일치하는 결과이며, 지리적으로 비교적 가까운 전주와 남원에서도 서로 다른 저항성 유전자에 반응하는 균계가 분포할 수 있음을 시사한다.
현재까지 도열병 저항성 관련 QTL은 500여개, 유전자는 100여개 가량 보고되었으며, 동정이 되어 특성이 상세히 밝혀진 유전자는 약 25개로(Li et al., 2019) 대부분이 NBS- LRR (nucleotide binding site leucine rich repeat) 단백질을 만들어 저항성 기작을 보인다(Jones & Dangl, 2006). qBL1.1의 물리적 위치(30,019,984~34,790,922 bp)는 도열병 저항성 유전자로 기존에 알려진 알려진 Pish/Pi35 유전자 영역(33,141,127-33,144,999 bp)을 포함하며, 2번 염색체의 qBL2.1 (34,000,118~35,242,550 bp)은 Pib 유전자 영역(35,108,842- 35,115,834bp)을 포함한다.
qBL1.1의 후보 유전자로 추정되는 pish/Pi35는 NBS-LRR단백질을 통한 저항성 기작을 보이며, 주변의 다른 NBS-LRR 유전자 3개와 나란히 위치해있다(Araki et al., 2003; Takahashi et al., 2010; Kitazawa et al., 2019). Pish/Pi35 (Os01g 0782100)는 동일 유전자의 서로 다른 대립인자로(Kitazawa et al., 2019), 일본 품종 Nipponbare가 보유한 것으로 알려진 Pish는 특정 균계에만 도열병 저항성을 갖는 반면 Hokkai188이 보유한 Pi35는 광범위 저항성을 보이는 점에서 차이가 있다(Nguyen et al., 2006; Takahashi et al., 2010). Pib유전자(Os02g0818450)는 동아시아 도열병 균계에 대하여 넓은 범위의 저항성을 부여하는 대표적인 도열병 저항성 유용 유전자로 알려져 있다(Wang et al.. 2001; Kitazawa et al., 2019). 추후 고시히카리와 백일미의 해당 유전자에 대한 염기서열 분석을 통하여 두 품종이 Pish/Pi35와 Pib에서 서로 다른 대립인자를 보유하고 있는지의 여부를 검토할 계획이다.
대부분의 도열병 저항성 유전자는 3~5년이 지나면 균계 진화에 의하여 저항성이 붕괴되는데(Devi et al., 2015), 국내 벼 품종이 보유한 도열병 저항성 유전자는 제한되어 있기에(Cho et al., 2004) 다양한 도열병 저항성 육종소재를 지속적으로 탐색하는 것은 매우 중요하다. 본 연구에서 탐지한 qBL1.1, qBL2.1은 추가 연구를 통하여 동정 및 정밀 분자표지 개발을 통해 도열병 저항성 분자육종에 유용하게 활용될 수 있을 것으로 예상한다.
KBRIL 394계통 중에서 고시히카리 또는 백일미와 농업형질이 유사하면서 도열병에 저항성을 보이는 계통을 아래와 같은 방법으로 선발하였다. KBRIL 394계통 중에서 출수기, 간장, 도열병 저항성을 기준으로 1) 출수기와 간장이 고시히카리와 유사하면서 도열병에 강한 계통(KR), 2) 출수기와 간장이 고시히카리와 유사하면서 도열병에 약한 계통(KS), 3) 출수기와 간장이 백일미와 유사하면서 도열병에 강한 계통(BR)을 각각 15계통씩 선발하여 그룹화하고, 출수기, 간장, 수장, 수수, 현미 천립중, 현미 장·폭, 현미 완전립률을 조사하였다. 3개의 그룹 중에서 고시히카리와 유사하면서 도열병에 강한(KR) 계통은 현미 완전립률이 다른 그룹에 비해 높은 것으로 관찰되었다. 추후 밥맛 검정, 수량성 등 추가적인 검정을 통하여 고시히카리의 우수한 밥맛을 지니면서 도열병 저항성을 보유한 고품질 밥쌀용 벼 품종 개발에 유용하게 활용될 것으로 기대된다. 또한 BR 그룹은 백일미와 유사하게 출수기가 빠르고 도열병에 강한 저항성을 보유하면서도 고시히카리 유사 그룹(KS)과 현미 외관에서 큰 차이를 보이지 않았으므로, 빠른 출수로 작부체계 상 다양한 돌려짓기에 유용한 고품질 조생종 품종 개발에 활용할 계획이다.
적 요
고시히카리는 도열병과 쓰러짐에 약하지만 밥맛 좋은 쌀로 유명하고, 육성된 지 60년이 넘은 지금까지도 일본에서 가장 많이 재배되는 품종이다. 고시히카리에 도열병에 강하면서 생육이 빠른 백일미를 교배한 RIL집단(KBRIL)에서 도열병 저항성에 대한 유전분석을 수행하여 저항성 유전자의 염색체 상 위치를 규명하고, 고시히카리의 우수한 미질을 보유하면서 도열병에도 강한 계통을 선발하기 위해 본 연구를 수행하였다. 주요 결과는 다음과 같다.
1. 고시히카리×백일미 RIL 394계통과 모·부본의 도열병 저항성(전주, 남원) 및 주요 농업형질을 조사하고, 유전분석을 위해 사용된 142계통으로 총 130개 SNP 마커, 1,272.7cM의 유전자지도를 작성하였다. 도열병 저항성 QTL 분석 결과 전주에서는 1번 염색체의 qBL1.1이, 남원에서는 전주와 동일한 qBL1.1과 추가로 2번 염색체의 qBL2.1이 탐지되었다.
2. RIL 394계통의 qBL1.1과 qBL2.1 유전자형을 도출하고 각 QTL의 백일미 대립인자 집적에 의한 도열병 저항성 강화 효과를 관찰하였다. 전주에서는 qBL1.1의 경우에만 백일미 대립인자 집적에 의하여 도열병 저항성이 강화되었다. 반면 남원에서는 qBL1.1, qBL2.1 모두 백일미 대립인자가 집적될 때 도열병 저항성이 강화되었다. qBL1.1, qBL2.1은 출수기, 간장, 수장, 수수를 포함한 주요 농업형질에는 영향을 미치지 않았다.
3. 고시히카리×백일미 RIL 394계통 중에서 출수기와 간장을 기준으로 고시히카리와 유사하면서 도열병에 약/강한(KS/KR) 계통과 백일미와 유사하면서 도열병에 강한(BR) 계통을 각 15계통씩 선발하였다. KR 그룹은 완전미율이 가장 우수하여 밥맛 검정, 수량성 등 추가조사를 통해 고시히카리의 우수한 밥맛을 지니면서 도열병 저항성을 보유한 고품질 밥쌀용 품종개발에 활용할 계획이다. 또한 BR그룹은 미질이 우수하면서 출수가 빠른 고품질 품종 개발에 유용할 것으로 기대된다.






