서 론
재료 및 방법
식물재료 및 특성조사
DNA 추출
멜론 유전자원의 유전적 구성 분석을 위한 PCR 수행
통계분석
멜론 유전자원의 유전적 구성 판단
결 과
멜론 유전자원 분포
멜론 유전자원의 형태적 특성
양적형질간 상관관계
멜론 질적형질별 양적형질 특성
주성분 분석
형태적 특성을 활용한 군집분석
멜론 유전자원의 유전적 구성
고 찰
적 요
서 론
멜론(Cucumis melo L.)은 다양한 국가에서 상업적으로 재배되는 중요한 작물로 우리나라에서도 국민 소득 향상과 건강 식품의 선호로 인하여 소비자들에게 인기가 높아지고 있다(Jung et al., 2015; Youn et al., 2011). 멜론은 우리나라를 비롯하여 태평양제도와 남미, 아시아, 아프리카, 오스트레일리아 등 전 대륙에 걸쳐 발견되는데 국가와 지역마다 재래종과 품종들이 혼재되어 재배되고 있다(Kerje & Grum, 2000). 특히, 타식성 작물로서 자연 교잡률이 높을 뿐만 아니라, 종간 혹은 아종간 교배로 인한 중간형 변종들이 존재하여 매우 높은 형태적 다양성을 보인다고 알려져 있다(Burger et al., 2009; Yi et al., 2004).
많은 유전자원센터들은 유전자원을 수집, 보존하고 그들 중 우량 육종 소재를 효율적으로 제공하는 역할을 하고 있다(Park et al., 2018). 수집된 자원은 그 기원과 특성에 따라 분류 관리하며 어떤 육종 목표에 적절한 재료로 활용할 수 있는지에 대한 정리 또한 중요하다(Nam et al., 2004). 농촌진흥청 국립농업과학원 농업유전자원센터는 신품종 개발, 신물질 탐색 및 생명공학 연구의 기본재료인 다양한 유전자원을 확보하고 있으며 수요자들에게 원활한 공급을 위해 종자량 확보 및 활력검정, 자원의 특성조사를 수행하고 있다(Kim et al., 2013).
유전자원의 특성 조사에는 수리분류학(Numerical taxonomy)의 발전에 따라 잎과 과실 및 화기 등에 대한 양적·질적 특성들은 물론 식물체내에 존재하는 페놀 화합물이나 당 등의 성분 조성의 차이, 처리 간의 생장 반응 등 다양한 형질들이 대상이 되고 있으며, 이러한 자료들에 기초하여 요인 분석, 주성분 분석과 군집분석 등의 여러 다변량 분석법이 적용되고 있다(Kim & Ko, 1995).
타식성 작물 육종에 있어서 다양한 유전자원의 수집, 특성평가뿐만 아니라 수집된 자원들의 유전적 다양성, 계통유연관계 및 집단 구조를 평가하는 것은 품종 개발을 위한 자식계통 육성 및 발굴, 그리고 교배조합 예측 등에 효율적으로 이용될 수 있다(Jang et al., 2013). 형태적 특성을 통한 다양성 분석은 1차적인 방법으로 여타 방법에 비해 비용 및 시간 소모가 적고 기술적 지식이 필요치 않으나 재배 환경에 영향을 받을 수 있어 정밀한 다른 방법을 사용하기 이전에 큰 범주의 그룹화를 위해 활용된다(Khalid et al., 2010; Malek et al., 2014). 이에 비해 분자마커를 이용한 분석은 환경의 영향을 받지 않으며 높은 재현성을 보여 형태적 분석보다 자원의 정밀한 다양성 분석에 이용될 수 있다(Smith et al., 1997).
농업유전자원센터에는 국내외에서 수집한 멜론 4834자원이 보존되고 있다. 본 연구에서는 멜론 유전자원의 육종소재 평가에 필요한 정보를 제공하고자 형태적, 지리적으로 다양하게 수집된 멜론 206자원을 대상으로 형태적 특성을 조사하고 그 결과를 바탕으로 다변량 분석을 통한 그들의 특성을 확인하였다. 또한 20개 simple sequence repeat (SSR) 마커를 이용하여 그들의 유전적 구성을 확인하고 육종소재로 활용가능한 자원 선별을 위한 기초자료를 확보하였다.
재료 및 방법
식물재료 및 특성조사
공시된 멜론(Cucumis melo L.) 206자원은 농촌진흥청 국립농업과학원 농업유전자원센터에 보존 중인 자원을 분양 받아 실험을 수행하였다(Table 1). 2018년 4월 20일에 농업유전자원센터 비닐 하우스 시설을 이용하여 자원당 4~6개체씩 주간 40 cm, 열간 200 cm 간격으로 농촌진흥청 표준시비법에 따라 재배하였다. 형질특성조사는 농업유전자원센터 멜론 특성조사기준을 참고하여 과실에 관한 7개의 특성(과장, 과폭, 과육 두께, 과형지수, 당도, 과피색, 과육색)과 개화일수, 성숙일수를 포함한 9개의 특성을 평가하였다(Table 2).
Table 1.
List of melon accessions used in this study.
| Acc. No. | Origin | Acc. No. | Origin | Acc. No. | Origin | Acc. No. | Origin | Acc. No. | Origin |
| K189601 | AFG* | K153014 | CZE | K189247 | IND | K190163 | MMR | K190020 | TUR |
| K189627 | AFG | K190669 | CZE | K189256 | IND | 907364 | MNG | K190415 | TUR |
| K189630 | AFG | K190857 | EGY | K189445 | IND | k014456 | MNG | K190416 | TUR |
| K189633 | AFG | K189159 | ESP | K189829 | IND | k014547 | MNG | K100483 | UKR |
| K189669 | AFG | K189210 | ESP | K190078 | IND | 807357 | PHL | K260967 | UKR |
| K189678 | AFG | K190335 | ESP | K190323 | IND | K100486 | RUS | K100482 | USA |
| k189685 | AFG | K190604 | ESP | K100480 | IRN | K261008 | RUS | K260944 | USA |
| K189686 | AFG | K190605 | ESP | K189769 | IRN | K261043 | RUS | K260947 | USA |
| K189690 | AFG | K190628 | ESP | K190190 | IRN | K261077 | RUS | K260949 | USA |
| K189698 | AFG | K190644 | ESP | K190197 | IRN | K190899 | SYR | K260951 | USA |
| K190236 | AFG | K190646 | ESP | K190313 | IRN | K190900 | SYR | K260954 | USA |
| K190283 | AFG | K190655 | ESP | K190332 | IRN | 805446 | TJK | K260957 | USA |
| K190292 | AFG | K190776 | ESP | K190346 | IRN | k003009 | TJK | K260994 | USA |
| K266894 | ARM | K190788 | ESP | K190349 | IRN | k003019 | TJK | K260995 | USA |
| K266912 | ARM | K190801 | ESP | K190439 | IRN | K247297 | TJK | 805354 | UZB |
| K266917 | ARM | K190806 | ESP | K190447 | IRN | K247726 | TJK | 805457 | UZB |
| K266920 | ARM | K190824 | ESP | K190691 | IRN | K247809 | TJK | 805504 | UZB |
| K266936 | ARM | K190833 | ESP | K190693 | IRN | K247831 | TJK | 805507 | UZB |
| K266940 | ARM | K190837 | ESP | K260979 | IRN | K264288 | TJK | 908855 | UZB |
| K266942 | ARM | K254420 | GEO | K190035 | IRQ | K264289 | TJK | 908861 | UZB |
| K267070 | ARM | K257488 | GEO | K190699 | ISR | k003004 | TKM | 908862 | UZB |
| K267102 | ARM | K257494 | GEO | K190884 | ISR | k003007 | TKM | k003008 | UZB |
| K267118 | ARM | K257496 | GEO | 803591 | JPN | k003013 | TKM | k003011 | UZB |
| K127079 | BGR | K257497 | GEO | 805440 | KAZ | K044199 | TKM | k003012 | UZB |
| K156332 | BGR | K257499 | GEO | 903224 | KAZ | K100474 | TKM | k003018 | UZB |
| K156378 | BGR | K257500 | GEO | k003015 | KAZ | K100478 | TKM | k003020 | UZB |
| K156600 | BGR | K261163 | GEO | K247306 | KAZ | K100481 | TKM | K014702 | UZB |
| k189746 | CAN | K261164 | GEO | K264285 | KAZ | k003014 | TUR | K044196 | UZB |
| K189748 | CAN | K261165 | GEO | k003010 | KGZ | K134068 | TUR | K044202 | UZB |
| K189757 | CAN | K266733 | GEO | K121370 | KGZ | K134120 | TUR | K100475 | UZB |
| k014348 | CHN | K266735 | GEO | K166774 | KOR | K134175 | TUR | K100479 | UZB |
| K051419 | CHN | K266738 | GEO | K166780 | KOR | K134188 | TUR | K100484 | UZB |
| K051446 | CHN | K266739 | GEO | K166782 | KOR | K134207 | TUR | K100488 | UZB |
| K051448 | CHN | K266740 | GEO | K166784 | KOR | K134279 | TUR | K260984 | UZB |
| K189116 | CHN | K268475 | GEO | K175600 | KOR | K145123 | TUR | K264291 | UZB |
| K189118 | CHN | K268477 | GEO | K175601 | KOR | K145188 | TUR | K226862 | VNM |
| K189480 | CHN | K268478 | GEO | K190742 | KOR | K145216 | TUR | K190710 | ZWE |
| K189809 | CHN | K190755 | HUN | K246072 | KOR | K189947 | TUR | K190721 | ZWE |
| K190944 | CHN | K190756 | HUN | K247642 | KOR | K189956 | TUR | ||
| K190949 | CHN | K018961 | IND | K252703 | KOR | K189970 | TUR | ||
| K190960 | CHN | K018962 | IND | K166351 | MDV | K189988 | TUR | ||
| K166368 | COL | K189237 | IND | K190690 | MEX | K190003 | TUR |
* AFG, Afghanistan.; ARM, Armenia; BGR, Bulgaria; CAN, Canada; CHN, China; COL, Colombia; CZE, Czech; EGY, Egypt; ESP, Spain; GEO, Georgia; HUN, Hungary; IND, India; IRN, Iran; IRQ, Iraq; ISR, Israel; JPN, Japan; KAZ, Kazakh-stan; KGZ, Kazkhstan; KOR, Korea; MDV, Maldives; MEX, Mexico; MMR, Myanmar; MNG, Mongolia; PHL, Philippines; RUS, Russia; SYR, Syria; TJK, Tajikstan; TKM, Turkmenistan; TUR, Turkey; UKR, Ukraine; USA, America; UZB, Uzbekistan; VNM, Vietnam; ZWE, Zimbabwe.
Table 2.
Descriptor for characterization of morphological traits of melon accessions.
DNA 추출
멜론 206자원의 Genomic DNA는 Qiagen DNA extraction kit (Qiagen, Hilden, Germany)를 사용하여 추출하였으며 추출 방법은 kit 내 사용방법을 따랐다. 추출된 DNA는 1% agarose gel에 전기영동, Spectrophotometry (Epoch, BioTek, Winooski, VT, USA)를 이용하여 DNA를 확인하고 최종 농도를 20 ng/ul로 맞춘 후 SSR마커 분석에 이용하였다.
멜론 유전자원의 유전적 구성 분석을 위한 PCR 수행
멜론 유전자원의 유전적 상태를 알아보기 위하여 Fukino et al. (2007)이 보고한 50개의 SSR 마커 중 PIC값이 0.5이상이며 PCR조건이 확립된 20개의 SSR 마커를 선발하였다(Table 3). PCR 반응은 Lee et al. (2019)의 방법을 따라 수행을 하였다. PCR 증폭 산물은 ABI prism 3500 DNA sequence (ABI3500, Thermo Fisher Scientific Inc., Wilmington, DE, USA)와 Gene Mapper Software (Version 4.0, Thermo Fisher Scientific Inc.)를 이용하여 분석하였다.
Table 3.
List of 20 SSR markers used in this study.
| Locus | F | R | PIC* |
| CMN04_01 | gaaaaggcgaaatgtttgaa | accaaaggaaaaagcggaag | 0.80 |
| CMN21_04 | gagctttgcccaaaaatcag | gaagcaggatgggttttgaa | 0.80 |
| CMN21_25 | tggaaactctgtgggtaggc | ttttgctgaaacgcaacaac | 0.80 |
| CMN09_76 | gacaattgatctggacagttttt | ccgtgatcaacaactattgaattt | 0.76 |
| CMN05_75 | gtggaccccactcgtaaaga | ggatgacggtcacaggaagt | 0.75 |
| CMN01_02 | cgatccaagttggcaagaat | actcttctcagccgcagatt | 0.71 |
| CMN04_03 | atcacagagaccgccaaaac | ggttgaagattgcgcttgat | 0.71 |
| CMN04_09 | tcttaacaactcccacgcct | ataggatctgccattgggc | 0.71 |
| CMN04_19 | ttcttcccaccaaacctacg | aaatggcagagagcgagaaa | 0.71 |
| CMN61_44 | tgttggagtttaatgaggaagga | agagaagatgaatggggcac | 0.78 |
| CMN22_54 | gtcgctacgtcagcctcttt | gacagggaagcaacgtaacc | 0.67 |
| CMN07_65 | cttagctgccattgcctttc | gaatcgcatatggtttcggt | 0.66 |
| CMN21_82 | attctggacgtcagattccg | tctccattcacgccttcttc | 0.66 |
| CMN05_17 | aggaaacacctttttgggga | aacccaggtcttgatgatgg | 0.65 |
| CMN53_36 | tgtggcttgatctatcgcag | cgtcggctagaggagaaatg | 0.59 |
| CMN05_89 | cttcatggctgggaagagaa | gtatggattgcctcctaccg | 0.57 |
| CMN07_70 | cctacaactcacgtgccctt | aaaccatcaaccaccgaaag | 0.54 |
| CMN08_79 | cgttcgcagatgtcaggtag | cttccgcacgaaacagattg | 0.54 |
| CMN01_35 | ttccgaaatctttgaacctctcca | cagagcagcaaaatccacaca | 0.52 |
| CMN01_38 | gctaaatatttgcaaactcgcc | agcttgacattccagaaggg | 0.51 |
통계분석
멜론 유전자원의 기술통계, 양적형질 간 상관관계, 주성분 분석은 PAST3 (Hammer et al., 2001)를 이용하여 분석을 하였으며 군집 분석 및 그룹 간 최소유의차 검정은 R 소프트웨어(http://www.r-project.org)에서 제공하는 패키지 agricolae, gplot2를 이용하여 분석하였다.
멜론 유전자원의 유전적 구성 판단
멜론 206자원의 유전적 구성에 대한 평가는 Lee et al. (2019)에서 제시한 방식에 따라 판단하였다(Table 4). 분석에 사용한 20개의 SSR 마커의 증폭 산물이 동일한 패턴을 보이는 경우는 근교계(Inbred) 또는 F1, 다른 패턴을 보일 경우에는 F2 이후의 분리세대 또는 종자 혼립의 4가지 형태로 자원을 구분하였다.
Table 4.
Prediction of genetic composition in 206 melon accessions.
결 과
멜론 유전자원 분포
본 연구에서 사용된 206개의 멜론 유전자원은 대륙별로 아시아 원산이 159자원(77.2%)으로 가장 많았으며 유럽 30자원(14.6%), 북아메리카 15자원(7.3%), 남아메리카와 아프리카 각 1자원(0.5%)씩 분포하였다(Fig. 1). 국가별로는 우즈베키스탄에서 수집한 자원이 21자원(10.2%)으로 가장 많았고 조지아, 터키에서 각각 18자원(8.7%), 스페인 16자원(7.8%) 순이었다(Table 1).
멜론 유전자원의 형태적 특성
멜론 유전자원의 형태적 특성은 개화일수, 성숙일수, 과장, 과폭, 과육 두께, 과형지수, 당도 등 7개의 양적 형질과 과피색과 과육색의 두가지 질적 형질을 조사하였다(Table 5).
Table 5.
Summary of seven agricultural traits in 206 melon accessions.
| Min | Max | Mean | SD | Median | Skewness | Kurtosis | CV (%) | F-value | |
| FD* | 57.5 | 86.0 | 65.2 | 6.0 | 63.8 | 1.0946 | 0.8213 | 9.2 | 5.468*** |
| MD | 15.0 | 58.0 | 42.3 | 6.5 | 42.3 | -0.2097 | 0.6305 | 15.5 | 5.061*** |
| FL | 8.0 | 29.3 | 17.7 | 4.6 | 17.4 | 0.2297 | -0.3449 | 26.3 | 8.538*** |
| FW | 6.4 | 17.2 | 11.5 | 2.0 | 11.5 | -0.1842 | 0.0526 | 17.3 | 4.953*** |
| L/W | 0.4 | 1.1 | 0.7 | 0.2 | 0.7 | 0.5718 | 0.1122 | 22.0 | 8.362*** |
| TH | 10.7 | 50.0 | 29.1 | 7.6 | 29.0 | -0.0801 | 0.0153 | 26.0 | 5.486*** |
| SU | 2.5 | 13.2 | 7.0 | 2.0 | 7.0 | 0.3517 | -0.1888 | 29.1 | 5.431*** |
개화일수는 57.5~86.0일로 분포하였으며 평균 65.2일로 나타났으며 성숙일수는 15.0~58일로 평균 42.3일로 분포하였다. 과장은 8.0~29.3 cm로 평균 17.7 cm, 과폭은 6.4~17.2 cm로 평균 11.5 cm를 보였으며 과형지수는 0.4~1.1로 평균 0.7로 나타났다. 과육의 두께는 10.7~50.0 mm로 평균 29.1 mm였으며 당도는 2.5~13.2 birx로 평균 7.0 brix를 보였다. 7개의 질적형질은 모두 자원간에 유의적 차이(p<0.001)를 보였으며 개화일수가 자원간 가장 낮은 변이계수(9.2%)를 보인 반면 당도가 가장 높은 변이계수(29.1%)를 보였다(Table 5).
과피색은 녹색 102자원(49%), 황색 90자원(44%), 백색 14자원(7%)으로 분포를 하였으며(Fig. 2A), 과육색은 흰색 115자원(56%), 황색 57자원(28%), 녹색 34자원(16%)으로 조사되었다(Fig. 2B).
양적형질간 상관관계
멜론 유전자원의 7가지 양적형질들의 상관관계는 Table 6에 나타냈다. 과장은 과폭(r=0.558, p<0.0001), 과육두께(r=5.14, p<0.001), 성숙일수(r=0.325, p<0.001)와 정의 상관을 보였으며 과형지수(r=-0.736, p<0.001)와는 부의 상관을 보였다. 과폭의 경우에는 과육두께(r=0.837, p<0.001)와 성숙일수(r=0.240, p<0.001)와 정의 상관을 보였다. 과육 두께는 당도(r=0.147, p<0.05)와 성숙일수(r=0.157, p<0.05)와 정의 상관을 보였다. 당도의 경우에는 개화기(r=-0.146, p<0.05)와는 부의 상관을 보인데 비해 성숙일수(r=0.269, p<0.01)와 과형지수(r=0.202, p<0.01)와는 정의 상관을 보였다. 개화일수는 성숙일수(r=-0.553, p<0.001)와 성숙일수는 과형지수(r=-0.205, p<0.001)와 각각 부의 상관을 보였다(Table 6).
Table 6.
Correlation among seven agricultural traits.
| FL* | FW | TH | SU | FD | MD | |
| FW | 0.558***** | |||||
| TH | 0.514*** | 0.837*** | ||||
| SU | -0.041 | 0.108 | 0.147* | |||
| FD | -0.086 | -0.0274 | 0.049 | -0.146* | ||
| MD | 0.325*** | 0.240*** | 0.157* | 0.269*** | -0.553*** | |
| L/W | -0.736*** | 0.0672 | -0.005 | 0.202** | 0.0165 | -0.205** |
멜론 질적형질별 양적형질 특성
멜론 유전자원 과육 및 과피색에 따른 양적형질 조사결과는 Table 7과 같다. 과피색은 과폭을 제외한 6개 양적형질과 모두 유의한 차이를 보였다. 과피색 중 흰색 과피를 보인 자원들이 가장 짧은 과장, 과육두께, 개화일수를 보였으며 가장 높은 당도와 과형지수를 보였다. 과육색의 경우 과피색과는 달리 과육 두께와 개화일수의 2개 양적형질에서만 유의한 차이를 나타냈으며 과육이 흰색인 자원이 가장 얇은 과육 두께와 짧은 개화일수를 보였다(Table 7).
Table 7.
Average for seven quantitative morphological traits of accessions per qualitative trait.
| Trait | FL* | FW | TH | SU | FD | MD | L/W | |
| Skin color | White | 12.8±4.4 c** | 10.7±2.4 ns | 25.6±9.6 b | 8.3±2.9 a | 62.3±5.8 b | 42.2±7.4 ab | 0.89±0.18 a |
| Green | 17.2±5.5 b | 11.4±2.7 | 28.6±9.9 a | 6.9±2.6 b | 64.3±7.3 a | 41.7±8.4 b | 0.69±0.16 b | |
| Yellow | 18.5±5.9 a | 11.4±2.5 | 29.2±9.6 a | 7.1±2.7 b | 64.6±7.3 a | 44.1±8.4 a | 0.66±0.20 c | |
| Flesh color | White | 17.1±6.0 ns | 11.2±2.6 ns | 28.1±9.6 b | 7.1±2.6 ns | 63.9±6.9 b | 43.0±8.2 ns | 0.70±0.20 ns |
| Yellow | 18.0±5.4 | 11.7±2.7 | 30.5±10.0 a | 7.2±2.8 | 65.9±8.0 a | 42.8±8.7 | 0.69±0.18 | |
| Green | 18.0±5.8 | 11.3±2.5 | 28.9±9.8 ab | 7.1±2.7 | 64.2±7.3 b | 43.3±8.4 | 0.67±0.19 | |
주성분 분석
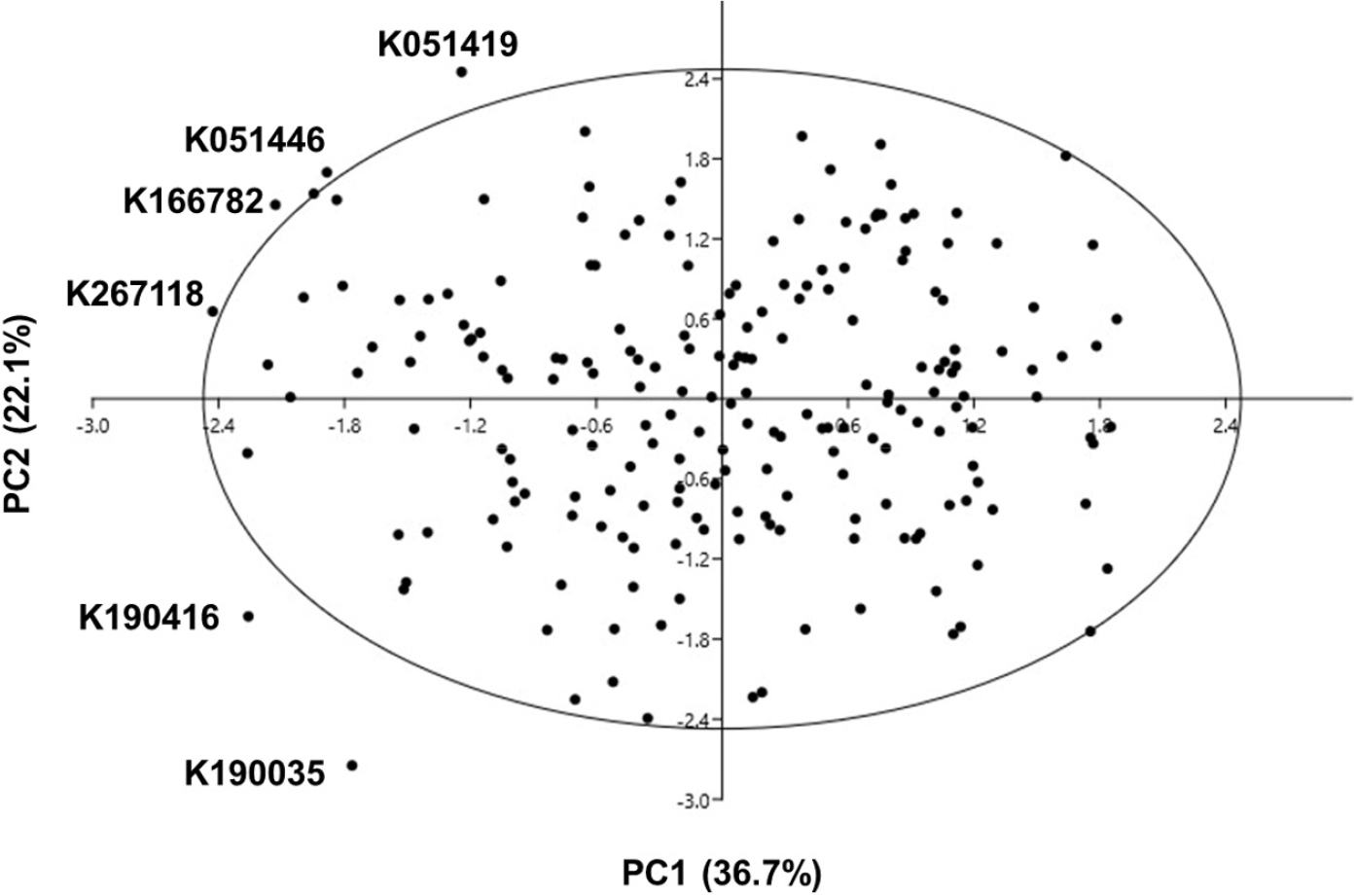
멜론 유전자원의 7개 양적 형질을 이용하여 주성분 분석을 수행한 결과는 Table 8과 같다. 주성분 분석을 통하여 얻어진 각 특성들에 대한 고유값(Eigen value)은 제1주성분(PC1)의 경우 2.569로 전체 분산에 대하여 36.7%의 기여도가 있는 것으로 분석되었으며, 고유값이 1이상인 제2주성분(PC2)과 제3주성분(PC3)의 고유 값은 각각 1.549와 1.517로 전체 분산에 대하여 80.5%의 높은 기여도를 나타내고 있다.
Table 8.
Eigen value, percentage of total variation, and component matrix for the principal component axes.
| Principal Components | PC 1 | PC 2 | PC 3 |
| Eigen value | 2.569 | 1.549 | 1.517 |
| % of Variance | 36.7 | 22.1 | 21.7 |
| Cumulative % | 36.7 | 58.8 | 80.5 |
| FL* | 0.538 | -0.243 | -0.261 |
| FW | 0.501 | -0.083 | 0.404 |
| TH | 0.484 | -0.140 | 0.417 |
| SU | 0.105 | 0.475 | 0.269 |
| FD | -0.157 | -0.595 | 0.264 |
| MD | 0.345 | 0.504 | -0.241 |
| L/W | -0.266 | 0.286 | 0.628 |
주성분 분석결과 고유값이 1이상으로 전체 분산에 대하여 80.5%의 기여도를 나타낸 제1주성분부터 제3주성분까지의 특성간 상관관계를 분석한 결과, 제1주성분은 -0.2226 (과형지수) ~ 0.538 (과장)로 상관계수가 분포를 하였으며 과폭, 과장, 과육두께 등의 요인에 높게 편중되어 있다. 제2주성분의 경우 -0.595 (개화일수) ~ 0.504 (성숙일수)로 상관계수가 분포를 하고 있으며, 개화일수와는 높은 부의 상관관계를 성숙일수와 당도와는 높은 정의 상관관계를 나타내고 있다. 제3주성분에서는 -0.241 (과장) ~ 0.628 (과형지수)로 상관계수가 분포를 하고 있으며 과형지수에서 가장 높은 상관계수를 나타냈다.
PCA 분석결과의 제1주성분과 제2주성분값을 2차원 공간상에 배열한 뒤 Hotelling T2 test를 이용하여 95%신뢰구간을 표시한 결과는 Fig. 3과 같다. 멜론 6자원(K051419, K051446, K166782, K267118, K190416, K190035)을 제외하고는 모두 범위 안에 들어오는 것을 확인할 수 있었다. 특이점을 보인 멜론 6자원 중 K051419, K166782, K051446, K267118은 짧은 과장을 보였으며 K190416, K190035의 경우에는 늦은 개화기를 보였다.
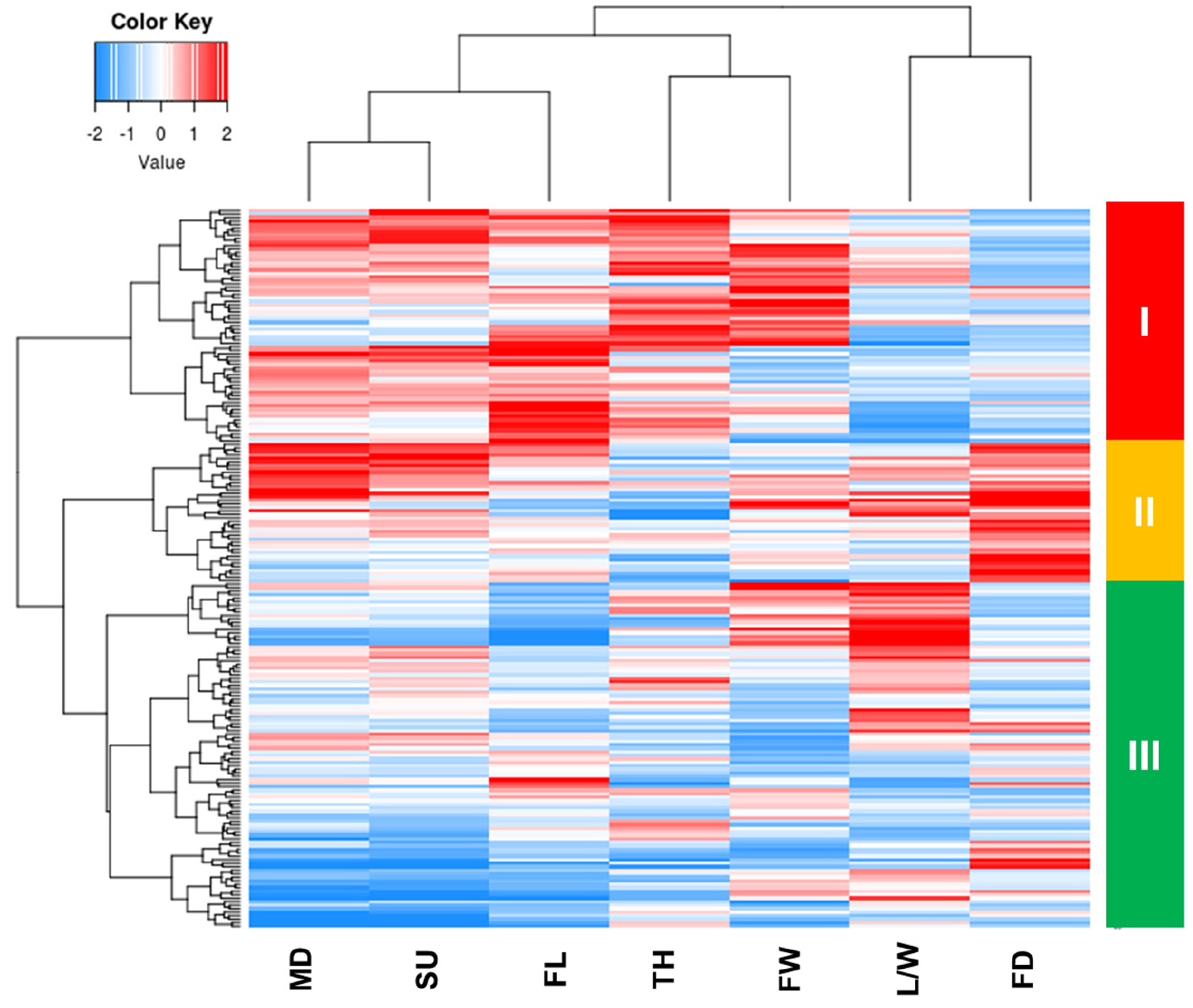
형태적 특성을 활용한 군집분석
206개 멜론 유전자원의 양적 형질을 이용하여 군집분석을 수행한 결과 3개의 그룹으로 나뉘었다(Fig. 4 and Table 9). 그룹 I, II, III은 각각 107자원, 43자원, 56자원으로 구성되었다. 그룹 I은 과장, 과폭, 과육두께가 다른 그룹보다 크며 당도도 높다. 또한 개화일수와 성숙일수도 다른 그룹보다 긴 특징을 보였다. 그룹 II의 경우 가장 짧은 과장을 보였으며 과형지수도 다른 그룹보다 높았다. 그룹 III의 경우 가장 좁은 과폭과 과육 두께를 보였다. 또한 가장 낮은 당도와 가장 짧은 성숙일수를 보였다.
Table 9.
Cluster average values for morphological traits of accessions per group.
| No. acc | FL* | FW | TH | SU | FD | MD | L/W | |
| I | 107 | 20.5±5.2 a | 12.5±2.3 a | 33.2±8.9 a | 7.8±2.7 a | 65.0±8.2 a | 45.4±8.5 a | 0.64±0.15 b |
| II | 43 | 13.2±3.7 c | 11.1±2.1 b | 26.4±7.3 b | 7.2±2.7 b | 62.9±5.2 b | 42.6±6.5 b | 0.88±0.20 a |
| III | 56 | 15.8±5.3 b | 9.6±2.5 c | 22.7±9.0 c | 6.0±2.2 c | 64.4±6.7 a | 39.5±8.1 c | 0.63±0.16 b |
멜론 유전자원의 유전적 구성
206개 멜론 유전자원의 유전적 상태는 Table 4와 같다. 그들 중 11자원이 동일한 마커패턴을 보였으며 195자원이 다른 패턴을 보였다. 동일한 패턴을 보인 11자원 중에서 K189118, K100486, K190292은 20개의 SSR 마커에서 모두 동형접합형 대립유전자(homozygote allele)를 보여 근교계(Inbred)로 예측할 수 있다. 반면 803591, 805507, 805446, K190605, K267118, K190960, K189480, K189669의 경우 1~3개의 마커에서 모두 이형접합형 대립유전자(heterozygote allele)를 보였으므로 이들은 F1일 것이라 예측할 수 있다. 마커 패턴이 다른 195자원 중 49자원은 마커당 두개 이하의 크기(size)및 대립유전자(allele)를 보였으나 그 패턴이 다양하였고 이를 근거로 F2세대 이후 분리 세대로 예측할 수 있었으며 146자원은 하나의 마커에 3개 이상의 크기(size) 또는 대립유전자(allele)이 확인 되어 혼립으로 예상하였다.
고 찰
멜론은 아프리카와 아시아가 원산지일 것이라 추정하지만 우리나라 참외를 포함하여 태평양 제도, 남미, 오스트레일리아 등 전세계에 걸쳐 발견된다(Kerje & Grum, 2000). 본 연구에서 사용된 멜론 자원들의 77.2%는 아시아에서 수집된 자원들이며 아프리카에서 수집된 자원은 1자원뿐이었다. 아시아 국가에 편중된 자원 수집은 대상국가 또는 국제기관에서 보유한 자원들 중 분양 가능한 자원들이 아시아 원산에 집중된 결과일 것이다. 또한 농업유전자원센터와 국제협력연구 등을 통해 자원의 수집, 도입이 활발했던 국가에서 도입된 자원이 다수를 차지한다. Lee et al. (2000)에 따르면 유전자원센터의 자원 구성은 62%가 국가와 국제기관으로부터 도입된 자원이라고 보고하였다. 본 연구에서 사용된 멜론 자원의 약 84.5% 또한 우즈베키스탄의 식물산업연구소(Uzbek Research Institute of Plant Industry), 미국 농무성 농업연구청(USDA-ARS), 러시아 바빌로프 연구소(Vavilov Research Institute) 등의 국가기관에서 도입이 되었으며 주로 재래종과 육성계통들로 구성되어 있다. 한편, 다른 대륙의 국가들과는 달리 아프리카 대륙에는 유전자원 관리기관을 운영하는 국가가 거의 없기 때문에 아프리카 자원의 분포가 적었을 것이라 생각된다(Mallor & Diaz, 2016).
멜론의 과실은 크기, 형태 색상 등 매우 다양하다고 보고되어 있다(Kirkbride, 1993; Nuñez-Palenius et al., 2008). 기존 보고에 따르면 과장이 4 cm에서 200 cm까지 다양한 크기를 보이며 무게도 50 g에서 15 kg까지 분포를 보인다고 하였다. 과형의 경우 소비자들의 선호도에 관여를 하는 형질로 멜론의 종류에 따라 다르지만 대체로 원형을 선호한다(농산업경영과, 2019). 본 연구에서도 멜론 206자원은 과장과 과폭, 과형지수 등이 다양하게 분포를 보였다 (Table 5). 특히 완전 구형(과형지수=1.0)인 8자원도 관찰되었다.
과육색에 있어서도 황색, 핑크색, 녹색, 흰색, 혼합색 등이 관찰되며, 과피색에서는 녹색, 황색, 백색, 오렌지색, 적색, 회색 등으로 다양한 자원이 관찰되었다는 보고가 있다(Goldman & Schrager, 2002; Kerje & Grum, 2000; Kirkbride, 1993). 과육색과 과피색의 경우 기존의 연구보고에서처럼 다양하지는 않으나 녹색, 황색, 백색의 자원들이 확인되었다. 멜론의 과육색은 엽록체와 carotenoid의 조합에 의해 결정되며 흰색, 녹색, 황색이 주요 색이다(Burger et al., 2009). Carotenoid 중에서 황색의 주 요소는 ß-carotene이며 ß-carotene의 함량에 따라 멜론의 과육색의 황색 정도가 결정된다(Nuñez-Palenius et al., 2008). 멜론의 ß-carotene은 비타민 A의 전구물질이며 중요한 항산화 성분으로 당근과 함께 프로비타민 A 섭취를 위한 식이 요소로 중요하다고 알려져 있다(Fleshman et al., 2011). 본 연구에서도 멜론 28자원이 과육색이 황색을 보였으며 이들의 ß-carotene 함량의 추가적 분석을 수행한다면 새로운 ß-carotene 함량을 목표로 하는 육종소재로 활용 가능할 것이다.
멜론의 품질을 결정하는 일차적인 기준은 당도이며 재배환경에 따라 차이를 보이나 2.1~14.8 brix를 보인다(Lee & Hyeon, 2003; Pitrat et al., 2000). 우리나라에서는 멜론의 종류에 따라 다르지만 12~13 brix이상의 과실이 유통되며 15 brix 이상의 멜론이 상품으로 인정되고 17~18 brix 이상이 초고품질 멜론으로 인정받고 있다(RDA, 2019). 일본의 경우 14 brix이상의 멜론이 상품으로 일본 시장에서 유통되고 있으며 15 brix이상의 품종들이 개발되어 있다(KFTC, 2018). (Burger et al., 2009)에 따르면 멜론의 당도가 최소 9brix 이상은 되어야 육종 소재로 육종가들이 활용할 수 있다고 하였다. 본 연구에서 멜론 206자원의 당도는 2.5~13.2 brix로 매우 다양한 분포를 보였으며 평균 7.0 brix로 낮은 편이었으나(Table 5) 이 중 33자원은 9 brix이상이었고 특히 803591의 경우 13.2 birx로 높은 당도를 나타내 새로운 육종소재로 이용 가능할 것이다.
기존에 우리는 수박 유전자원을 이용하여 그들의 유전적 구성을 SSR 마커를 이용하여 확인하고 그들 중 근교계(Inbred)로 예측되는 6자원을 보고하고 그들의 활용 방안에 대해 토의하였다(Lee et al., 2019). 본 연구에서는 20개의 SSR마커 결과를 토대로 유전적 구성을 예측하고 이중 근교계로 예측되는 멜론 3자원을 확인 할 수 있었다. 20개 마커는 12개의 멜론 염색체에 골고루 분포하며 높은 PIC값을 보여 유전적 구성 예측에 사용하기 적합하였다. 특히 근교계(Inbred)의 경우 20개의 마커가 모두 동형접합형 대립유전자(homozygote allele)의 결과를 보여야 하며 이는 매우 낮은 확률로 존재하기 때문에 유전적 구성 예측에 충분한 판단근거가 될 것이다. 타식성 작물의 유전자원 활용을 위해서는 재래종, 육성계통 등 다양한 집단을 수집하여 유용한 유전자 및 대립유전자를 보존하는 것이 매우 중요하다. 이와 더불어 수집한 자원의 유전적 상태를 확인하여 근교계, F1, 혼립 등을 구분하여 육종소재로 이용할 수 있도록 준비해야 한다. 타식성 작물의 육종에는 다양한 자원의 수집과 보존, 평가뿐만 아니라 우수한 자식계통을 얻기 위한 유전적 고정이 필요하다(Jang et al., 2013) 본 연구에서 근교계로 예측된 3자원은 멜론 육종 프로그램에 직접 활용할 경우 자식계통 육성 및 발굴을 위한 과정 없이 육종 연한을 단축할 수 있을 것이라 생각된다.
멜론은 2014년 이후 재배와 소비가 감소하는 추세를 보이고 있으나 국내에서는 아직도 선물용 고급 과일로 형태, 무게, 당도 등이 복합병저항성과 함께 중요한 육종 목표이다(RDA, 2019). 본 연구에서는 다양한 국가에서 수집된 멜론 206자원을 육종 소재로 활용하기 위하여 소비자가 선호하는 과형과 연관된 형질 - 과장, 과폭, 과육두께 - 및 당도를 중점으로 자원을 평가하였다. 본 연구의 평가 결과 형태 및 당도가 우수하여 멜론의 품질 향상에 도움이 될 수 있는 자원들을 확인 할 수 있었다. 또한 다양한 과육색을 갖는 자원들을 확인하여 기능성 멜론에 대한 기초 정보도 확보하였다. 이들 특성정보와 유전적 구성 분석 결과는 새로운 육종소재 개발에 유용할 것이며 추후, ß-carotene등을 포함한 기능성 물질 함량 및 복합병저항성 검정, 멜론 재배 적합 관련 특성 등의 분석이 필요할 것이다.
적 요
농업유전자원센터에 보존 중인 멜론(Cucumis melo L.) 206자원에 대한 형태적 특성을 분석하였다. 형태적 특성은 과장, 과폭, 과형지수, 과육두께, 당도, 개화일수, 성숙일수 등 7가지 양적형질과 과피색, 과육색 등 2가지 질적형질을 조사하고 다변량분석을 하였다. 또한 20개 SSR 마커를 이용하여 그들의 유전적 구성을 분석하였다.
1. 멜론 유전자원 중 아시아 국가에서 수집한 자원이 77.2%로 가장 많았으며 국가별로는 우즈베키스탄에서 수집한 자원이 가장 많았다.
2. 7가지 양적형질중에서 개화일수가 자원간 가장 적은 변이(CV=9.2%)를 보였으며 당도(CV=29.1%)가 가장 높은 변이를 보였다.
3. 질적 형질 중 과피색은 녹색(102자원, 49%), 과육색은 흰색(115자원, 56%)이 가장 많았다.
4. 주성분 분석 결과 제1주성분(36.7%), 제2주성분(22.1%), 제3주성분(21.7%)이 전체 변량의 80.5%를 나타내었다.
5. 군집분석 결과 멜론 206자원은 3개의 그룹으로 나뉘었으며 각각 107자원, 43자원, 56자원으로 구성이 되었다. 이 중 그룹 I에 속한 자원들이 다른 그룹의 자원들 보다 과장, 과폭이 길고 당도가 높은 특징을 보였다.
6. SSR 마커를 이용한 유전적 상태 구분 결과 K189118, K100486, K190292은 모두 동형접합형 대립유전자(Homozygote allele)를 보였으며 이를 토대로 근교계(Inbred) 자원으로 예측 할 수 있었다.
본 연구 결과는 멜론 과일 품질 향상을 위한 육종 프로그램에 기초자료로 활용될 수 있을 것이다.